4. 图像的 NumPy 速成课程#
scikit-image 中的图像由 NumPy ndarray 表示。因此,可以使用标准的 NumPy 方法来操作数组,从而实现许多常见的操作
>>> import skimage as ski
>>> camera = ski.data.camera()
>>> type(camera)
<type 'numpy.ndarray'>
注意
诸如 pandas.DataFrame 或 xarray.DataArray 之类的标记数组类数据类型在 scikit-image 中不受原生支持。但是,在某些假设下,存储在这些类型中的数据可以转换为 numpy.ndarray(请参阅 pandas.DataFrame.to_numpy() 和 xarray.DataArray.data)。特别是,这些转换会忽略采样坐标(DataFrame.index、DataFrame.columns 或 DataArray.coords),这可能会导致数据表示不准确,例如,当原始数据点间隔不均匀时。
检索图像的几何信息和像素数量
>>> camera.shape
(512, 512)
>>> camera.size
262144
检索有关图像强度值的统计信息
>>> camera.min(), camera.max()
(0, 255)
>>> camera.mean()
118.31400299072266
表示图像的 NumPy 数组可以是不同的整数或浮点数值类型。有关这些类型以及 scikit-image 如何处理它们的更多信息,请参阅 图像数据类型及其含义。
4.1. NumPy 索引#
NumPy 索引既可用于查看像素值,也可用于修改它们
>>> # Get the value of the pixel at the 10th row and 20th column
>>> camera[10, 20]
153
>>> # Set to black the pixel at the 3rd row and 10th column
>>> camera[3, 10] = 0
注意!在 NumPy 索引中,第一个维度(camera.shape[0])对应于行,而第二个维度(camera.shape[1])对应于列,原点(camera[0, 0])位于左上角。这与矩阵/线性代数表示法相匹配,但与笛卡尔 (x, y) 坐标相反。有关更多详细信息,请参阅下面的坐标约定。
除了单个像素之外,还可以使用 NumPy 的不同索引功能来访问/修改整组像素的值。
切片
>>> # Set the first ten lines to "black" (0)
>>> camera[:10] = 0
掩码(使用布尔掩码进行索引)
>>> mask = camera < 87
>>> # Set to "white" (255) the pixels where mask is True
>>> camera[mask] = 255
花式索引(使用索引集进行索引)
>>> import numpy as np
>>> inds_r = np.arange(len(camera))
>>> inds_c = 4 * inds_r % len(camera)
>>> camera[inds_r, inds_c] = 0
当您需要选择一组像素来执行操作时,掩码非常有用。掩码可以是与图像形状相同的任何布尔数组(或可以广播到图像形状的形状)。这可用于定义感兴趣区域,例如,一个圆盘
>>> nrows, ncols = camera.shape
>>> row, col = np.ogrid[:nrows, :ncols]
>>> cnt_row, cnt_col = nrows / 2, ncols / 2
>>> outer_disk_mask = ((row - cnt_row)**2 + (col - cnt_col)**2 >
... (nrows / 2)**2)
>>> camera[outer_disk_mask] = 0

NumPy 的布尔运算可用于定义更复杂的掩码
>>> lower_half = row > cnt_row
>>> lower_half_disk = np.logical_and(lower_half, outer_disk_mask)
>>> camera = data.camera()
>>> camera[lower_half_disk] = 0
4.2. 彩色图像#
以上所有内容对于彩色图像仍然适用。彩色图像是一个 NumPy 数组,带有用于通道的额外尾部维度
>>> cat = ski.data.chelsea()
>>> type(cat)
<type 'numpy.ndarray'>
>>> cat.shape
(300, 451, 3)
这表明 cat 是一个 300 x 451 像素的图像,带有三个通道(红色、绿色和蓝色)。和之前一样,我们可以获取和设置像素值
>>> cat[10, 20]
array([151, 129, 115], dtype=uint8)
>>> # Set the pixel at (50th row, 60th column) to "black"
>>> cat[50, 60] = 0
>>> # set the pixel at (50th row, 61st column) to "green"
>>> cat[50, 61] = [0, 255, 0] # [red, green, blue]
我们还可以像对上面的灰度图像一样,对 2D 多通道图像使用 2D 布尔掩码
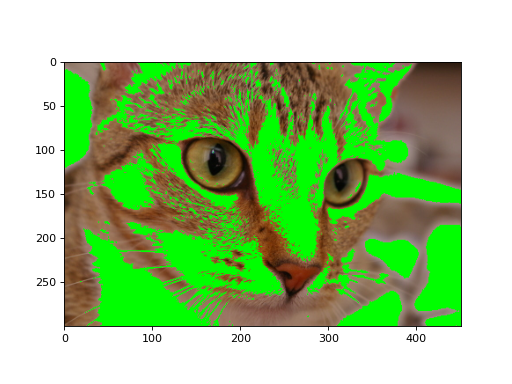
在 2D 彩色图像上使用 2D 掩码#
skimage.data 中包含的示例彩色图像的通道存储在最后一个轴上,尽管其他软件可能遵循不同的约定。支持彩色图像的 scikit-image 库函数具有一个 channel_axis 参数,该参数可用于指定数组的哪个轴对应于通道。
4.3. 坐标约定#
由于 scikit-image 使用 NumPy 数组表示图像,因此坐标约定必须匹配。二维 (2D) 灰度图像(如上面的 camera)通过行和列(缩写为 (row, col) 或 (r, c))进行索引,左上角的最低元素为 (0, 0)。在库的各个部分,您还会看到 rr 和 cc 指的是行和列坐标的列表。我们将此约定与 (x, y) 区分开来,后者通常表示标准的笛卡尔坐标,其中 x 是水平坐标,y 是垂直坐标,原点在左下角(例如,Matplotlib 轴使用此约定)。
对于多通道图像,任何维度(数组轴)都可以用于颜色通道,并用 channel 或 ch 表示。在 scikit-image 0.19 之前,此通道维度始终是最后一个,但在当前版本中,可以通过 channel_axis 参数指定通道维度。需要多通道数据的函数默认为 channel_axis=-1。否则,函数默认为 channel_axis=None,表示不假定任何轴对应于通道。
最后,对于体积 (3D) 图像,如视频、磁共振成像 (MRI) 扫描、共聚焦显微镜等,我们将前导维度称为 plane,缩写为 pln 或 p。
这些约定总结如下
图像类型 |
坐标 |
|---|---|
2D 灰度 |
(row, col) |
2D 多通道(例如,RGB) |
(row, col, ch) |
3D 灰度 |
(pln, row, col) |
3D 多通道 |
(pln, row, col, ch) |
请注意,ch 的位置由 channel_axis 参数控制。
scikit-image 中的许多函数可以直接对 3D 图像进行操作
>>> import numpy as np
>>> import scipy as sp
>>> import skimage as ski
>>> rng = np.random.default_rng()
>>> im3d = rng.random((100, 1000, 1000))
>>> seeds = sp.ndimage.label(im3d < 0.1)[0]
>>> ws = ski.segmentation.watershed(im3d, seeds)
但是,在许多情况下,第三个空间维度的分辨率低于其他两个。一些 scikit-image 函数提供了一个 spacing 关键字参数来帮助处理此类数据
>>> slics = ski.segmentation.slic(im3d, spacing=[5, 1, 1], channel_axis=None)
有时,必须按平面进行处理。当平面沿前导维度堆叠时(与我们的约定一致),可以使用以下语法
>>> edges = np.empty_like(im3d)
>>> for pln, image in enumerate(im3d):
... # Iterate over the leading dimension
... edges[pln] = ski.filters.sobel(image)
4.4. 关于数组维度顺序的说明#
尽管轴的标记可能看起来是任意的,但它会对操作速度产生重大影响。这是因为现代处理器从不只从内存中检索一个项目,而是检索一整块相邻的项目(称为预取操作)。因此,在内存中彼此相邻的元素的处理速度快于它们分散时的处理速度,即使操作数量相同
>>> def in_order_multiply(arr, scalar):
... for plane in list(range(arr.shape[0])):
... arr[plane, :, :] *= scalar
...
>>> def out_of_order_multiply(arr, scalar):
... for plane in list(range(arr.shape[2])):
... arr[:, :, plane] *= scalar
...
>>> import time
>>> rng = np.random.default_rng()
>>> im3d = rng.random((100, 1024, 1024))
>>> t0 = time.time(); x = in_order_multiply(im3d, 5); t1 = time.time()
>>> print("%.2f seconds" % (t1 - t0))
0.14 seconds
>>> s0 = time.time(); x = out_of_order_multiply(im3d, 5); s1 = time.time()
>>> print("%.2f seconds" % (s1 - s0))
1.18 seconds
>>> print("Speedup: %.1fx" % ((s1 - s0) / (t1 - t0)))
Speedup: 8.6x
当最后一个/最右边的维度变得更大时,加速效果会更加明显。在开发算法时,值得考虑数据局部性。特别是,scikit-image 默认使用 C 连续数组。当使用嵌套循环时,数组的最后一个/最右边的维度应该在计算的最内层循环中。在上面的示例中,*= numpy 运算符会迭代所有剩余的维度。
4.5. 关于时间维度的说明#
尽管 scikit-image 目前不提供专门处理时变 3D 数据的功能,但它与 NumPy 数组的兼容性使我们能够非常自然地处理形状为 (t, pln, row, col, ch) 的 5D 数组
>>> for timepoint in image5d:
... # Each timepoint is a 3D multichannel image
... do_something_with(timepoint)
我们可以按如下方式补充上表
图像类型 |
坐标 |
|---|---|
2D 彩色视频 |
(t, 行, 列, 通道) |
3D 彩色视频 |
(t, 平面, 行, 列, 通道) |
