skimage.metrics#
对应于图像的度量标准,例如,距离度量、相似度等。
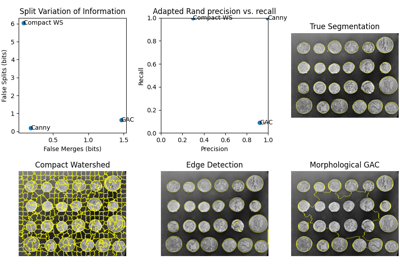
计算 SNEMI3D 竞赛定义的自适应兰德误差。 |
|
返回匹配分割中所有区域的列联表。 |
|
计算给定图像的非零元素之间的豪斯多夫距离。 |
|
返回给定图像的非零元素之间相隔豪斯多夫距离的点对。 |
|
计算两个图像之间的均方误差。 |
|
计算归一化互信息 (NMI)。 |
|
计算两个图像之间的归一化均方根误差 (NRMSE)。 |
|
计算图像的峰值信噪比 (PSNR)。 |
|
计算两个图像之间的平均结构相似性指数。 |
|
返回与 VI 相关的对称条件熵。 |
- skimage.metrics.adapted_rand_error(image_true=None, image_test=None, *, table=None, ignore_labels=(0,), alpha=0.5)[源代码]#
计算 SNEMI3D 竞赛定义的自适应兰德误差。 [1]
- 参数:
- image_trueint 类型的 ndarray
真值标签图像,与 im_test 形状相同。
- image_testint 类型的 ndarray
测试图像。
- tablescipy.sparse 以 crs 格式表示的数组,可选
使用 skimage.evaluate.contingency_table 构建的列联表。如果为 None,则会动态计算。
- ignore_labelsint 类型的序列,可选
要忽略的标签。真实图像中标记有这些值中的任何一个的部分都不会计入分数。
- alphafloat,可选
在自适应兰德误差计算中赋予精确率和召回率的相对权重。
- 返回:
- arefloat
自适应兰德误差。
- precfloat
自适应兰德精确率:这是在测试标签图像和真实图像中具有相同标签的像素对的数量,除以测试图像中的数量。
- recfloat
自适应兰德召回率:这是在测试标签图像和真实图像中具有相同标签的像素对的数量,除以真实图像中的数量。
注释
在真实分割中标签为 0 的像素在分数中会被忽略。
自适应兰德误差的计算公式如下
\(1 - \frac{\sum_{ij} p_{ij}^{2}}{\alpha \sum_{k} s_{k}^{2} + (1-\alpha)\sum_{k} t_{k}^{2}}\),其中 \(p_{ij}\) 是像素在测试图像和真实图像中具有相同标签的概率,\(t_{k}\) 是像素在真实图像中具有标签 \(k\) 的概率,\(s_{k}\) 是像素在测试图像中具有标签 \(k\) 的概率。
默认行为是在自适应兰德误差计算中平均权重精确率和召回率。当 alpha = 0 时,自适应兰德误差 = 召回率。当 alpha = 1 时,自适应兰德误差 = 精确率。
参考文献
[1]Arganda-Carreras I, Turaga SC, Berger DR 等。(2015) 众包创建用于连接组学的图像分割算法。Front. Neuroanat. 9:142. DOI:10.3389/fnana.2015.00142
- skimage.metrics.contingency_table(im_true, im_test, *, ignore_labels=None, normalize=False, sparse_type='matrix')[源代码]#
返回匹配分割中所有区域的列联表。
- 参数:
- im_trueint 类型的 ndarray
真值标签图像,与 im_test 形状相同。
- im_testint 类型的 ndarray
测试图像。
- ignore_labelsint 类型的序列,可选
要忽略的标签。真实图像中标记有这些值中的任何一个的部分都不会计入分数。
- normalizebool
确定是否按像素数归一化列联表。
- sparse_type{“matrix”, “array”},可选
cont的返回类型,可以是scipy.sparse.csr_array或scipy.sparse.csr_matrix(默认)。
- 返回:
- contscipy.sparse.csr_matrix 或 scipy.sparse.csr_array
列联表。
cont[i, j]将等于在im_true中标记为i且在im_test中标记为j的体素数量。根据sparse_type,这可以作为scipy.sparse.csr_array返回。
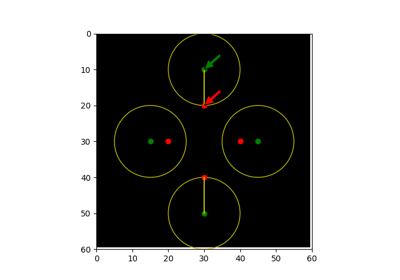
- skimage.metrics.hausdorff_distance(image0, image1, method='standard')[源代码]#
计算给定图像的非零元素之间的豪斯多夫距离。
- 参数:
- image0, image1ndarray
True表示包含在一组点中的点的数组。两个数组必须具有相同的形状。- method{‘standard’, ‘modified’},可选,默认 = ‘standard’
用于计算豪斯多夫距离的方法。
standard是标准的豪斯多夫距离,而modified是修正的豪斯多夫距离。
- 返回:
- distancefloat
使用欧几里得距离,
image0和image1中非零像素的坐标之间的豪斯多夫距离。
注释
豪斯多夫距离 [1] 是
image0上的任意点与其在image1上的最近点之间的最大距离,反之亦然。在 Dubuisson 等人的以下工作中,已证明修正的豪斯多夫距离 (MHD) 比有向豪斯多夫距离 (HD) 表现更好 [2]。该函数计算正向和反向平均距离并返回两者中较大的一个。参考文献
[2]M. P. Dubuisson 和 A. K. Jain。用于对象匹配的修正的豪斯多夫距离。ICPR94 中,第 A:566-568 页,以色列耶路撒冷,1994 年。 DOI:10.1109/ICPR.1994.576361 http://citeseerx.ist.psu.edu/viewdoc/summary?doi=10.1.1.1.8155
示例
>>> points_a = (3, 0) >>> points_b = (6, 0) >>> shape = (7, 1) >>> image_a = np.zeros(shape, dtype=bool) >>> image_b = np.zeros(shape, dtype=bool) >>> image_a[points_a] = True >>> image_b[points_b] = True >>> hausdorff_distance(image_a, image_b) 3.0
- skimage.metrics.hausdorff_pair(image0, image1)[源代码]#
返回给定图像的非零元素之间相隔豪斯多夫距离的点对。
豪斯多夫距离 [1] 是
image0上的任意点与其在image1上的最近点之间的最大距离,反之亦然。- 参数:
- image0, image1ndarray
True表示包含在一组点中的点的数组。两个数组必须具有相同的形状。
- 返回:
- point_a, point_b数组
一对点,它们之间具有豪斯多夫距离。
参考文献
示例
>>> points_a = (3, 0) >>> points_b = (6, 0) >>> shape = (7, 1) >>> image_a = np.zeros(shape, dtype=bool) >>> image_b = np.zeros(shape, dtype=bool) >>> image_a[points_a] = True >>> image_b[points_b] = True >>> hausdorff_pair(image_a, image_b) (array([3, 0]), array([6, 0]))
- skimage.metrics.mean_squared_error(image0, image1)[源代码]#
计算两个图像之间的均方误差。
- 参数:
- image0, image1ndarray
图像。任意维度,必须具有相同的形状。
- 返回:
- mse浮点数
均方误差 (MSE) 指标。
注释
在 0.16 版本中更改:此函数已从
skimage.measure.compare_mse重命名为skimage.metrics.mean_squared_error。
- skimage.metrics.normalized_mutual_information(image0, image1, *, bins=100)[源代码]#
计算归一化互信息 (NMI)。
\(A\) 和 \(B\) 的归一化互信息由下式给出
.. math::
Y(A, B) = frac{H(A) + H(B)}{H(A, B)}
其中 \(H(X) := - \sum_{x \in X}{x \log x}\) 是熵。
Colin Studholme 及其同事提出,它在图像配准中很有用 [1]。它的范围从 1(完全不相关的图像值)到 2(完全相关的图像值,无论是正相关还是负相关)。
- 参数:
- image0, image1ndarray
要比较的图像。两个输入图像必须具有相同的维度数。
- binsint 或 int 序列,可选
联合直方图的每个轴上的 bin 数量。
- 返回:
- nmi浮点数
两个数组之间的归一化互信息,以
bins给定的粒度计算。较高的 NMI 表示输入图像更相似。
- 引发:
- ValueError
如果图像的维度数不同。
注释
如果两个输入图像的形状不同,则较小的图像会用零填充。
参考文献
[1]C. Studholme, D.L.G. Hill, & D.J. Hawkes (1999). An overlap invariant entropy measure of 3D medical image alignment. Pattern Recognition 32(1):71-86 DOI:10.1016/S0031-3203(98)00091-0
- skimage.metrics.normalized_root_mse(image_true, image_test, *, normalization='euclidean')[源代码]#
计算两个图像之间的归一化均方根误差 (NRMSE)。
- 参数:
- image_truendarray
真值图像,与 im_test 形状相同。
- image_testndarray
测试图像。
- normalization{‘euclidean’, ‘min-max’, ‘mean’},可选
控制在 NRMSE 分母中使用的归一化方法。文献中没有标准的归一化方法 [1]。此处可用的方法如下
‘euclidean’:通过
im_true的平均欧几里德范数进行归一化NRMSE = RMSE * sqrt(N) / || im_true ||
其中 || . || 表示 Frobenius 范数,
N = im_true.size。此结果等效于NRMSE = || im_true - im_test || / || im_true ||.
‘min-max’:通过
im_true的强度范围进行归一化。‘mean’:通过
im_true的平均值进行归一化
- 返回:
- nrmse浮点数
NRMSE 指标。
注释
在 0.16 版本中更改:此函数已从
skimage.measure.compare_nrmse重命名为skimage.metrics.normalized_root_mse。参考文献
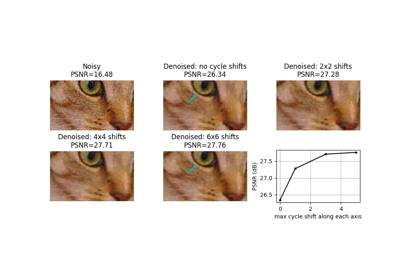
- skimage.metrics.peak_signal_noise_ratio(image_true, image_test, *, data_range=None)[源代码]#
计算图像的峰值信噪比 (PSNR)。
- 参数:
- image_truendarray
真值图像,与 im_test 形状相同。
- image_testndarray
测试图像。
- data_rangeint,可选
输入图像的数据范围(最小值和最大值之间的距离)。默认情况下,这是从图像数据类型估计的。
- 返回:
- psnr浮点数
PSNR 指标。
注释
在 0.16 版本中更改:此函数已从
skimage.measure.compare_psnr重命名为skimage.metrics.peak_signal_noise_ratio。参考文献
- skimage.metrics.structural_similarity(im1, im2, *, win_size=None, gradient=False, data_range=None, channel_axis=None, gaussian_weights=False, full=False, **kwargs)[源代码]#
计算两个图像之间的平均结构相似性指数。请注意浮点图像的
data_range参数。- 参数:
- im1, im2ndarray
图像。任意维度,形状相同。
- win_sizeint 或 None,可选
比较中使用的滑动窗口的边长。必须是奇数值。如果
gaussian_weights为 True,则忽略此值,窗口大小将取决于sigma。- gradientbool,可选
如果为 True,则还返回相对于 im2 的梯度。
- data_rangefloat,可选
输入图像的数据范围(最大值和最小值之间的差值)。默认情况下,这是从图像数据类型估计的。对于浮点图像数据,此估计可能不正确。因此,建议始终显式传递此标量值(请参见下面的注释)。
- channel_axisint 或 None,可选
如果为 None,则假定图像为灰度(单通道)图像。否则,此参数指示数组的哪个轴对应于通道。
在 0.19 版本中添加:
channel_axis在 0.19 中添加。- gaussian_weightsbool,可选
如果为 True,则每个补丁的均值和方差将通过宽度为 sigma=1.5 的归一化高斯核进行空间加权。
- fullbool,可选
如果为 True,则还返回完整的结构相似性图像。
- 返回:
- mssim浮点数
图像上的平均结构相似性指数。
- gradndarray
im1 和 im2 之间的结构相似性的梯度 [2]。仅当
gradient设置为 True 时才会返回此值。- Sndarray
完整的 SSIM 图像。仅当
full设置为 True 时才会返回此值。
- 其他参数:
注释
如果未指定
data_range,则根据图像数据类型自动猜测范围。但是,对于浮点图像数据,此估计会产生所需范围值的两倍,因为skimage.util.dtype.py中的dtype_range定义了 -1 到 +1 的间隔。这会产生 2 的估计值,而不是在使用图像数据时最常需要的 1(因为负光强度是无意义的)。在使用类似 YCbCr 的颜色数据时,请注意每个通道的范围都不同(Cb 和 Cr 的范围是 Y 的两倍),因此不能通过一次调用此函数来计算通道平均 SSIM,因为假设每个通道的范围相同。为了匹配 Wang 等人的实现 [1],请将
gaussian_weights设置为 True,将sigma设置为 1.5,将use_sample_covariance设置为 False,并指定data_range参数。在 0.16 版本中更改:此函数已从
skimage.measure.compare_ssim重命名为skimage.metrics.structural_similarity。参考文献
[1] (1,2,3)Wang, Z., Bovik, A. C., Sheikh, H. R., & Simoncelli, E. P. (2004). Image quality assessment: From error visibility to structural similarity. IEEE Transactions on Image Processing, 13, 600-612. https://ece.uwaterloo.ca/~z70wang/publications/ssim.pdf, DOI:10.1109/TIP.2003.819861
[2]Avanaki, A. N. (2009). Exact global histogram specification optimized for structural similarity. Optical Review, 16, 613-621. arXiv:0901.0065 DOI:10.1007/s10043-009-0119-z
- skimage.metrics.variation_of_information(image0=None, image1=None, *, table=None, ignore_labels=())[source]#
返回与 VI 相关的对称条件熵。 [1]
信息的变异定义为 VI(X,Y) = H(X|Y) + H(Y|X)。 如果 X 是真实分割,那么 H(X|Y) 可以解释为欠分割量,H(Y|X) 可以解释为过分割量。 换句话说,完美的过分割将有 H(X|Y)=0,而完美的欠分割将有 H(Y|X)=0。
- 参数:
- image0, image1int 类型的 ndarray
标签图像/分割,必须具有相同的形状。
- tablescipy.sparse 中 csr 格式的数组,可选
使用 skimage.evaluate.contingency_table 构建的列联表。 如果为 None,则将使用 skimage.evaluate.contingency_table 计算。 如果给定,则将从此表计算熵,并且将忽略任何图像。
- ignore_labelsint 类型的序列,可选
要忽略的标签。真实图像中标记有这些值中的任何一个的部分都不会计入分数。
- 返回:
- vifloat 类型的 ndarray,形状 (2,)
image1|image0 和 image0|image1 的条件熵。
参考文献
[1]Marina Meilă (2007), 比较聚类——基于信息距离, Journal of Multivariate Analysis, 第 98 卷,第 5 期,第 873-895 页, ISSN 0047-259X, DOI:10.1016/j.jmva.2006.11.013.