skimage.graph#
基于图的操作,例如,最短路径。
这包括创建图像中像素的邻接图、查找图像中的中心像素、查找跨像素的(最小成本)路径、合并和切割图等。
查找具有最高接近中心性的像素。 |
|
对区域邻接图执行归一化图切割。 |
|
合并权重小于阈值的分隔区域。 |
|
执行 RAG 的分层合并。 |
|
创建图像中像素的邻接图。 |
|
根据区域边界计算 RAG |
|
使用平均颜色计算区域邻接图。 |
|
如何使用 MCP 和 MCP_Geometric 类的简单示例。 |
|
查找通过 n 维数组从一侧到另一侧的最短路径。 |
|
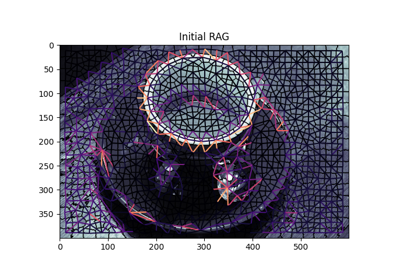
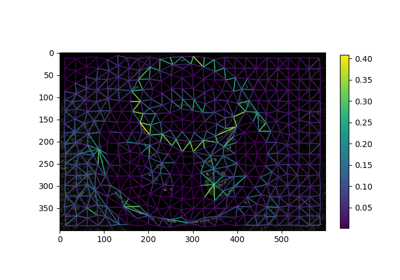
在图像上显示区域邻接图。 |
|
一个用于查找给定 n 维成本数组中的最小成本路径的类。 |
|
使用距离加权最小成本函数连接源点。 |
|
查找通过 N 维成本数组的最小成本路径。 |
|
查找通过 n 维成本数组的距离加权最小成本路径。 |
|
图像的区域邻接图 (RAG),子类 |
- skimage.graph.central_pixel(graph, nodes=None, shape=None, partition_size=100)[源代码]#
查找具有最高接近中心性的像素。
接近中心性是节点到每个其他节点的最短距离总和的倒数。
- 参数:
- graphscipy.sparse.csr_array 或 scipy.sparse.csr_matrix
图的稀疏表示。
- nodes整数数组
图像中图中每个节点的扁平索引。如果未提供,则返回的值将是输入图中的索引。
- shape整数元组
嵌入节点的图像的形状。如果提供,则返回的坐标是与输入形状具有相同维度的 NumPy 多索引。否则,返回的坐标是在
nodes中提供的扁平索引。- partition_size整数
此函数计算图中每对节点之间的最短路径距离。这可能会导致非常大的 (N*N) 矩阵。作为简单的性能调整,距离值以
partition_size的批量计算,从而导致仅 partition_size*N 的内存需求。
- 返回:
- position整数或整数元组
如果给定形状,则为图像中中心像素的坐标。否则,该像素的扁平索引。
- distances浮点数数组
从每个节点到每个其他可达节点的距离总和。
- skimage.graph.cut_normalized(labels, rag, thresh=0.001, num_cuts=10, in_place=True, max_edge=1.0, *, rng=None)[源代码]#
对区域邻接图执行归一化图切割。
给定图像的标签及其相似度 RAG,对其递归执行双向归一化切割。属于无法进一步切割的子图的所有节点都会在输出中分配一个唯一的标签。
- 参数:
- labelsndarray
标签数组。
- ragRAG
区域邻接图。
- thresh浮点数
阈值。如果 N-cut 的值超过
thresh,则不会进一步细分子图。- num_cuts整数
在确定最佳值之前要执行的 N-cut 的数量。
- in_place布尔值
如果设置,则就地修改
rag。对于每个节点n,函数将设置一个新属性rag.nodes[n]['ncut label']。- max_edge浮点数,可选
RAG 中边的最大可能值。这对应于相同区域之间的边。这用于将自边放入 RAG 中。
- rng{
numpy.random.Generator, int}, 可选 伪随机数生成器。默认情况下,使用 PCG64 生成器(请参阅
numpy.random.default_rng())。如果rng是一个整数,则用于为生成器设定种子。rng用于确定scipy.sparse.linalg.eigsh的起始点。
- 返回:
- outndarray
新的标记数组。
参考文献
[1]Shi, J.; Malik, J., “归一化切割和图像分割”,模式分析和机器智能,IEEE Transactions on, vol. 22, no. 8, pp. 888-905, 2000 年 8 月。
示例
>>> from skimage import data, segmentation, graph >>> img = data.astronaut() >>> labels = segmentation.slic(img) >>> rag = graph.rag_mean_color(img, labels, mode='similarity') >>> new_labels = graph.cut_normalized(labels, rag)
- skimage.graph.cut_threshold(labels, rag, thresh, in_place=True)[源代码]#
合并权重小于阈值的分隔区域。
给定图像的标签及其 RAG,通过组合其节点之间的权重小于给定阈值的区域来输出新标签。
- 参数:
- labelsndarray
标签数组。
- ragRAG
区域邻接图。
- thresh浮点数
阈值。连接权重较小的边的区域将组合在一起。
- in_place布尔值
如果设置,则就地修改
rag。该函数将删除权重小于thresh的边。如果设置为False,则该函数在继续之前会复制rag。
- 返回:
- outndarray
新的标记数组。
参考文献
[1]Alain Tremeau 和 Philippe Colantoni “应用于彩色图像分割的区域邻接图” DOI:10.1109/83.841950
示例
>>> from skimage import data, segmentation, graph >>> img = data.astronaut() >>> labels = segmentation.slic(img) >>> rag = graph.rag_mean_color(img, labels) >>> new_labels = graph.cut_threshold(labels, rag, 10)
- skimage.graph.merge_hierarchical(labels, rag, thresh, rag_copy, in_place_merge, merge_func, weight_func)[源代码]#
执行 RAG 的分层合并。
贪婪地合并最相似的一对节点,直到没有低于
thresh的边保留。- 参数:
- labelsndarray
标签数组。
- ragRAG
区域邻接图。
- thresh浮点数
权重小于
thresh的边连接的区域会被合并。- rag_copybool
如果设置,则在修改之前复制 RAG。
- in_place_mergebool
如果设置,则节点将就地合并。否则,每次合并都会创建一个新节点。
- merge_funccallable
此函数在合并两个节点之前调用。对于 RAG
graph,在合并src和dst时,其调用方式如下:merge_func(graph, src, dst)。- weight_funccallable
用于计算与合并节点相邻的新节点权重的函数。此函数直接作为
weight_func参数提供给merge_nodes。
- 返回:
- outndarray
新的标记数组。
- skimage.graph.pixel_graph(image, *, mask=None, edge_function=None, connectivity=1, spacing=None, sparse_type='matrix')[source]#
创建图像中像素的邻接图。
mask 为 True 的像素是返回图中的节点,并且它们根据连通性参数通过边连接到它们的邻居。默认情况下,当给出 mask 时,或者当图像本身是 mask 时,边的值是像素之间的欧几里得距离。
但是,如果给出一个没有 mask 的 int 或 float 值图像,则边的值是相邻像素之间强度差的绝对值,并乘以欧几里得距离。
- 参数:
- imagearray
输入图像。如果图像类型为 bool,则它也将用作 mask。
- maskbool 数组
使用哪些像素。如果为 None,则使用整个图像的图。
- edge_functioncallable
一个函数,接收像素值数组、相邻像素值数组和距离数组,并返回边的值。如果没有给出函数,则边的值只是距离。
- connectivityint
像素邻域的平方连通性:允许考虑像素为邻居的正交步数。 有关详细信息,请参阅
scipy.ndimage.generate_binary_structure。- spacingfloat 元组
沿每个轴的像素间距。
- sparse_type{“matrix”, “array”}, 可选
graph的返回类型,可以是scipy.sparse.csr_array或scipy.sparse.csr_matrix(默认)。
- 返回:
- graphscipy.sparse.csr_matrix 或 scipy.sparse.csr_array
稀疏邻接矩阵,其中条目 (i, j) 如果节点 i 和 j 是邻居,则为 1,否则为 0。根据
sparse_type,这可以作为scipy.sparse.csr_array返回。- nodes整数数组
图的节点。这些对应于 mask 中非零像素的扁平索引。
- skimage.graph.rag_boundary(labels, edge_map, connectivity=2)[source]#
根据区域边界计算 RAG
给定图像的初始分割及其边缘图,此方法构造相应的区域邻接图 (RAG)。RAG 中的每个节点表示图像中一组具有相同标签的像素,标签位于
labels中。两个相邻区域之间的权重是edge_map中沿其边界的平均值。- labelsndarray
标记的图像。
- edge_mapndarray
这应该与
labels的形状相同。对于沿两个相邻区域之间边界的所有像素,edge_map中相应像素的平均值是它们之间的边权重。- connectivityint,可选
彼此之间的平方距离小于
connectivity的像素被认为是相邻的。它的范围可以从 1 到labels.ndim。它的行为与scipy.ndimage.generate_binary_structure中的connectivity参数相同。
示例
>>> from skimage import data, segmentation, filters, color, graph >>> img = data.chelsea() >>> labels = segmentation.slic(img) >>> edge_map = filters.sobel(color.rgb2gray(img)) >>> rag = graph.rag_boundary(labels, edge_map)
- skimage.graph.rag_mean_color(image, labels, connectivity=2, mode='distance', sigma=255.0)[source]#
使用平均颜色计算区域邻接图。
给定图像及其初始分割,此方法构造相应的区域邻接图 (RAG)。RAG 中的每个节点表示
image中一组具有相同标签的像素,标签位于labels中。两个相邻区域之间的权重表示两个区域的相似或不相似程度,具体取决于mode参数。- 参数:
- imagendarray,形状 (M, N[, …, P], 3)
输入图像。
- labelsndarray,形状 (M, N[, …, P])
标记的图像。这应该比
image少一维。如果image的维度为(M, N, 3),则labels的维度应为(M, N)。- connectivityint,可选
彼此之间的平方距离小于
connectivity的像素被认为是相邻的。它的范围可以从 1 到labels.ndim。它的行为与scipy.ndimage.generate_binary_structure中的connectivity参数相同。- mode{‘distance’, ‘similarity’}, 可选
分配边权重的策略。
‘distance’:两个相邻区域之间的权重是 \(|c_1 - c_2|\),其中 \(c_1\) 和 \(c_2\) 是两个区域的平均颜色。它表示它们平均颜色中的欧几里得距离。
‘similarity’:两个相邻区域之间的权重是 \(e^{-d^2/sigma}\),其中 \(d=|c_1 - c_2|\),其中 \(c_1\) 和 \(c_2\) 是两个区域的平均颜色。它表示两个区域的相似程度。
- sigmafloat,可选
当
mode为 “similarity” 时用于计算。它控制两个颜色之间的接近程度,以便它们对应的边权重具有意义。 一个非常大的sigma值可能使任何两种颜色都表现得好像它们是相似的。
- 返回:
- outRAG
区域邻接图。
参考文献
[1]Alain Tremeau 和 Philippe Colantoni “应用于彩色图像分割的区域邻接图” DOI:10.1109/83.841950
示例
>>> from skimage import data, segmentation, graph >>> img = data.astronaut() >>> labels = segmentation.slic(img) >>> rag = graph.rag_mean_color(img, labels)
- skimage.graph.route_through_array(array, start, end, fully_connected=True, geometric=True)[源代码]#
如何使用 MCP 和 MCP_Geometric 类的简单示例。
有关路径查找算法的解释,请参阅 MCP 和 MCP_Geometric 类的文档。
- 参数:
- 返回:
- pathlist
定义从
start到end的路径的 n 维索引元组列表。- costfloat
路径的成本。如果
geometric为 False,则路径的成本是沿路径的array值之和。 如果geometric为 True,则进行更精细的计算(请参阅 MCP_Geometric 类的文档)。
另请参阅
示例
>>> import numpy as np >>> from skimage.graph import route_through_array >>> >>> image = np.array([[1, 3], [10, 12]]) >>> image array([[ 1, 3], [10, 12]]) >>> # Forbid diagonal steps >>> route_through_array(image, [0, 0], [1, 1], fully_connected=False) ([(0, 0), (0, 1), (1, 1)], 9.5) >>> # Now allow diagonal steps: the path goes directly from start to end >>> route_through_array(image, [0, 0], [1, 1]) ([(0, 0), (1, 1)], 9.19238815542512) >>> # Cost is the sum of array values along the path (16 = 1 + 3 + 12) >>> route_through_array(image, [0, 0], [1, 1], fully_connected=False, ... geometric=False) ([(0, 0), (0, 1), (1, 1)], 16.0) >>> # Larger array where we display the path that is selected >>> image = np.arange((36)).reshape((6, 6)) >>> image array([[ 0, 1, 2, 3, 4, 5], [ 6, 7, 8, 9, 10, 11], [12, 13, 14, 15, 16, 17], [18, 19, 20, 21, 22, 23], [24, 25, 26, 27, 28, 29], [30, 31, 32, 33, 34, 35]]) >>> # Find the path with lowest cost >>> indices, weight = route_through_array(image, (0, 0), (5, 5)) >>> indices = np.stack(indices, axis=-1) >>> path = np.zeros_like(image) >>> path[indices[0], indices[1]] = 1 >>> path array([[1, 1, 1, 1, 1, 0], [0, 0, 0, 0, 0, 1], [0, 0, 0, 0, 0, 1], [0, 0, 0, 0, 0, 1], [0, 0, 0, 0, 0, 1], [0, 0, 0, 0, 0, 1]])
- skimage.graph.shortest_path(arr, reach=1, axis=-1, output_indexlist=False)[源代码]#
查找通过 n 维数组从一侧到另一侧的最短路径。
- 参数:
- arrfloat64 的 ndarray
- reachint,可选
默认情况下(
reach = 1),最短路径对于其向前移动的每一步只能向上或向下移动一行(即,路径梯度限制为 1)。reach定义了每一步可以沿每个非轴维度跳过的元素数量。- axisint,可选
路径必须始终向前移动的轴(默认为 -1)
- output_indexlistbool,可选
有关说明,请参阅返回值
p。
- 返回:
- pint 的 iterable
对于沿
axis的每一步,最短路径的坐标。 如果output_indexlist为 True,则路径将作为索引到arr的 n 维元组列表返回。 如果为 False,则路径将作为数组返回,该数组列出沿轴维度每一步的非轴维度的路径坐标。 也就是说,p.shape == (arr.shape[axis], arr.ndim-1),只是在返回之前挤压 p,因此如果arr.ndim == 2,则p.shape == (arr.shape[axis],)- costfloat
路径的成本。 这是沿路径的所有差异的绝对和。
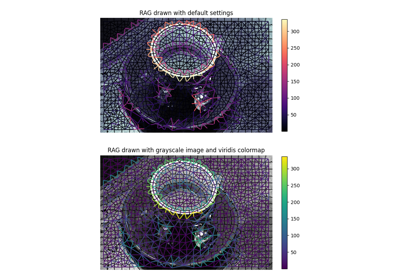
- skimage.graph.show_rag(labels, rag, image, border_color='black', edge_width=1.5, edge_cmap='magma', img_cmap='bone', in_place=True, ax=None)[源代码]#
在图像上显示区域邻接图。
给定一个标记的图像及其对应的 RAG,在图像上显示 RAG 的节点和边,并使用指定的颜色。 边显示在图像中 2 个相邻区域的质心之间。
- 参数:
- labelsndarray,形状 (M, N)
标记的图像。
- ragRAG
区域邻接图。
- imagendarray,形状 (M, N[, 3])
输入图像。 如果
colormap为None,则图像应为 RGB 格式。- border_color颜色规格,可选
绘制区域之间边界的颜色。
- edge_widthfloat,可选
绘制 RAG 边的粗细。
- edge_cmap
matplotlib.colors.Colormap,可选 用于绘制边的任何 matplotlib 颜色图。
- img_cmap
matplotlib.colors.Colormap,可选 用于绘制图像的任何 matplotlib 颜色图。 如果设置为
None,则图像按原样绘制。- in_placebool,可选
如果设置,则 RAG 将就地修改。 对于每个节点
n,该函数将设置一个新属性rag.nodes[n]['centroid']。- ax
matplotlib.axes.Axes,可选 要在其上绘制的轴。 如果未指定,则会创建新轴并在其上绘制。
- 返回:
- lc
matplotlib.collections.LineCollection 表示图的边的线集合。 可以将其传递给
matplotlib.figure.Figure.colorbar()函数。
- lc
示例
>>> from skimage import data, segmentation, graph >>> import matplotlib.pyplot as plt >>> >>> img = data.coffee() >>> labels = segmentation.slic(img) >>> g = graph.rag_mean_color(img, labels) >>> lc = graph.show_rag(labels, g, img) >>> cbar = plt.colorbar(lc)
- class skimage.graph.MCP(costs, offsets=None, fully_connected=True, sampling=None)#
基类:
object一个用于查找给定 n 维成本数组中的最小成本路径的类。
给定一个 n 维成本数组,可以使用此类查找通过该数组从任何一组点到任何另一组点的最小成本路径。 基本用法是初始化类并使用一个或多个起始索引(以及可选的结束索引列表)调用 find_costs()。 之后,多次调用 traceback() 以查找从任何给定结束位置到最近的起始索引的路径。 可以通过重复调用 find_costs() 来找到通过同一成本数组的新路径。
路径的成本计算方式很简单,即为路径上每个点的
costs数组值的总和。另一方面,MCP_Geometric 类考虑了对角线移动和轴向移动的长度不同这一事实,并相应地对路径成本进行加权。具有无限或负成本的数组元素将被忽略,累积成本溢出为无限的路径也将被忽略。
- 参数:
- costsndarray
- offsetsiterable, optional
偏移元组列表:每个偏移指定从给定 n 维位置进行的有效移动。如果未提供,则将使用 make_offsets() 和
fully_connected参数值构造对应于单连接或全连接 n 维邻域的偏移。- fully_connectedbool, optional
如果未提供
offsets,则此参数确定生成的邻域的连通性。如果为 true,则路径可以在costs数组元素之间沿对角线移动;否则只允许轴向移动。- samplingtuple, optional
对于每个维度,指定两个单元格/体素之间的距离。如果未给出或为 None,则假定距离为单位距离。
- 属性:
- __init__(costs, offsets=None, fully_connected=True, sampling=None)#
请参阅类文档。
- find_costs(starts, ends=None, find_all_ends=True, max_coverage=1.0, max_cumulative_cost=None, max_cost=None)#
查找从给定起始点到指定结束点的最小成本路径。
此方法查找从任何一个指定的起始索引到指定结束索引的最小成本路径。如果未给出结束位置,则会找到到 costs 数组中每个位置的最小成本路径。
- 参数:
- startsiterable
n 维起始索引列表(其中 n 是
costs数组的维度)。将找到到最近/最便宜的起始点的最小成本路径。- endsiterable, optional
n 维结束索引列表。
- find_all_endsbool, optional
如果为“True”(默认),则会找到到每个指定结束位置的最小成本路径;否则,当找到到任何结束位置的路径时,算法将停止。(如果未指定
ends,则此参数不起作用。)
- 返回:
- cumulative_costsndarray
与
costs数组的形状相同;此数组记录从最近/最便宜的起始索引到每个被考虑的索引的最小成本路径。(如果指定了ends,则并非数组中的所有元素都会被考虑:未评估的位置的累积成本将为 inf。如果find_all_ends为“False”,则只有一个指定的结束位置具有有限的累积成本。)- tracebackndarray
与
costs数组的形状相同;此数组包含从其前任索引到任何给定索引的偏移量。偏移索引索引到offsets属性,这是一个 n 维偏移数组。在 2 维情况下,如果 offsets[traceback[x, y]] 是 (-1, -1),则表示最小成本路径中 [x, y] 的前任是 [x+1, y+1]。请注意,如果 offset_index 是 -1,则表示未考虑给定索引。
- goal_reached(index, cumcost)#
int goal_reached(int index, float cumcost) 在从堆中弹出一个索引后,在检查邻居之前,每次迭代都会调用此方法。
可以重载此方法以修改 MCP 算法的行为。一个示例可能是在达到某个累积成本或前沿距离种子点一定距离时停止算法。
如果算法不应检查当前点的邻居,则此方法应返回 1;如果算法现在已完成,则应返回 2。
- offsets#
- traceback(end)#
通过预先计算的回溯数组追踪最小成本路径。
此便捷函数从提供给 find_costs() 的起始索引之一重建到给定结束位置的最小成本路径,find_costs() 必须事先调用。在 find_costs() 运行后,可以根据需要多次调用此函数。
- 参数:
- enditerable
一个到
costs数组的 n 维索引。
- 返回:
- tracebackn 维元组列表
一个到
costs数组的索引列表,从传递给 find_costs() 的起始位置之一开始,到给定的end索引结束。这些索引指定从任何给定起始索引到end索引的最小成本路径。(该路径的总成本可以从 find_costs() 返回的cumulative_costs数组中读取。)
- class skimage.graph.MCP_Connect(costs, offsets=None, fully_connected=True)#
基类:
MCP使用距离加权最小成本函数连接源点。
前沿从每个种子点同时增长,同时跟踪前沿的起源。当两个前沿相遇时,会调用 create_connection()。必须重载此方法,以使用适合于应用程序的方式处理找到的边缘。
- __init__(*args, **kwargs)#
- create_connection(id1, id2, tb1, tb2, cost1, cost2)#
create_connection id1, id2, pos1, pos2, cost1, cost2)
重载此方法以跟踪在 MCP 处理期间发现的连接。请注意,具有相同 id 的连接可能会被多次找到(但位置和成本不同)。
在调用此方法时,两个点都被“冻结”,MCP算法不会再次访问它们。
- 参数:
- id1int
第一个邻居的起始种子点 ID。
- id2int
第二个邻居的起始种子点 ID。
- pos1tuple
连接中第一个邻居的索引。
- pos2tuple
连接中第二个邻居的索引。
- cost1float
pos1处的累积成本。- cost2float
pos2处的累积成本。
- find_costs(starts, ends=None, find_all_ends=True, max_coverage=1.0, max_cumulative_cost=None, max_cost=None)#
查找从给定起始点到指定结束点的最小成本路径。
此方法查找从任何一个指定的起始索引到指定结束索引的最小成本路径。如果未给出结束位置,则会找到到 costs 数组中每个位置的最小成本路径。
- 参数:
- startsiterable
n 维起始索引列表(其中 n 是
costs数组的维度)。将找到到最近/最便宜的起始点的最小成本路径。- endsiterable, optional
n 维结束索引列表。
- find_all_endsbool, optional
如果为“True”(默认),则会找到到每个指定结束位置的最小成本路径;否则,当找到到任何结束位置的路径时,算法将停止。(如果未指定
ends,则此参数不起作用。)
- 返回:
- cumulative_costsndarray
与
costs数组的形状相同;此数组记录从最近/最便宜的起始索引到每个被考虑的索引的最小成本路径。(如果指定了ends,则并非数组中的所有元素都会被考虑:未评估的位置的累积成本将为 inf。如果find_all_ends为“False”,则只有一个指定的结束位置具有有限的累积成本。)- tracebackndarray
与
costs数组形状相同;此数组包含从其前驱索引到任何给定索引的偏移量。偏移量索引是offsets属性的索引,该属性是一个 n 维偏移数组。在二维情况下,如果 offsets[traceback[x, y]] 为 (-1, -1),则表示 [x, y] 在到达某个起始位置的最小成本路径中的前驱是 [x+1, y+1]。请注意,如果 offset_index 为 -1,则表示未考虑给定的索引。
- goal_reached(index, cumcost)#
int goal_reached(int index, float cumcost) 在从堆中弹出一个索引后,在检查邻居之前,每次迭代都会调用此方法。
可以重载此方法以修改 MCP 算法的行为。一个示例可能是在达到某个累积成本或前沿距离种子点一定距离时停止算法。
如果算法不应检查当前点的邻居,则此方法应返回 1;如果算法现在已完成,则应返回 2。
- offsets#
- traceback(end)#
通过预先计算的回溯数组追踪最小成本路径。
此便捷函数从提供给 find_costs() 的起始索引之一重建到给定结束位置的最小成本路径,find_costs() 必须事先调用。在 find_costs() 运行后,可以根据需要多次调用此函数。
- 参数:
- enditerable
一个到
costs数组的 n 维索引。
- 返回:
- tracebackn 维元组列表
一个到
costs数组的索引列表,从传递给 find_costs() 的起始位置之一开始,到给定的end索引结束。这些索引指定从任何给定起始索引到end索引的最小成本路径。(该路径的总成本可以从 find_costs() 返回的cumulative_costs数组中读取。)
- class skimage.graph.MCP_Flexible(costs, offsets=None, fully_connected=True)#
基类:
MCP查找通过 N 维成本数组的最小成本路径。
有关完整详细信息,请参阅 MCP 的文档。此类与 MCP 的区别在于,可以重载几个方法(来自纯 Python)以修改算法的行为和/或基于 MCP 创建自定义算法。请注意,goal_reached 也可以在 MCP 类中重载。
- __init__(costs, offsets=None, fully_connected=True, sampling=None)#
请参阅类文档。
- examine_neighbor(index, new_index, offset_length)#
一旦两个节点都被冻结,就会为每对相邻节点调用此方法。
可以重载此方法以获取有关相邻节点的信息,和/或修改 MCP 算法的行为。一个例子是 MCP_Connect 类,它使用此钩子检查相遇前沿。
- find_costs(starts, ends=None, find_all_ends=True, max_coverage=1.0, max_cumulative_cost=None, max_cost=None)#
查找从给定起始点到指定结束点的最小成本路径。
此方法查找从任何一个指定的起始索引到指定结束索引的最小成本路径。如果未给出结束位置,则会找到到 costs 数组中每个位置的最小成本路径。
- 参数:
- startsiterable
n 维起始索引列表(其中 n 是
costs数组的维度)。将找到到最近/最便宜的起始点的最小成本路径。- endsiterable, optional
n 维结束索引列表。
- find_all_endsbool, optional
如果为“True”(默认),则会找到到每个指定结束位置的最小成本路径;否则,当找到到任何结束位置的路径时,算法将停止。(如果未指定
ends,则此参数不起作用。)
- 返回:
- cumulative_costsndarray
与
costs数组的形状相同;此数组记录从最近/最便宜的起始索引到每个被考虑的索引的最小成本路径。(如果指定了ends,则并非数组中的所有元素都会被考虑:未评估的位置的累积成本将为 inf。如果find_all_ends为“False”,则只有一个指定的结束位置具有有限的累积成本。)- tracebackndarray
与
costs数组形状相同;此数组包含从其前驱索引到任何给定索引的偏移量。偏移量索引是offsets属性的索引,该属性是一个 n 维偏移数组。在二维情况下,如果 offsets[traceback[x, y]] 为 (-1, -1),则表示 [x, y] 在到达某个起始位置的最小成本路径中的前驱是 [x+1, y+1]。请注意,如果 offset_index 为 -1,则表示未考虑给定的索引。
- goal_reached(index, cumcost)#
int goal_reached(int index, float cumcost) 在从堆中弹出一个索引后,在检查邻居之前,每次迭代都会调用此方法。
可以重载此方法以修改 MCP 算法的行为。一个示例可能是在达到某个累积成本或前沿距离种子点一定距离时停止算法。
如果算法不应检查当前点的邻居,则此方法应返回 1;如果算法现在已完成,则应返回 2。
- offsets#
- traceback(end)#
通过预先计算的回溯数组追踪最小成本路径。
此便捷函数从提供给 find_costs() 的起始索引之一重建到给定结束位置的最小成本路径,find_costs() 必须事先调用。在 find_costs() 运行后,可以根据需要多次调用此函数。
- 参数:
- enditerable
一个到
costs数组的 n 维索引。
- 返回:
- tracebackn 维元组列表
一个到
costs数组的索引列表,从传递给 find_costs() 的起始位置之一开始,到给定的end索引结束。这些索引指定从任何给定起始索引到end索引的最小成本路径。(该路径的总成本可以从 find_costs() 返回的cumulative_costs数组中读取。)
- travel_cost(old_cost, new_cost, offset_length)#
此方法计算从当前节点到下一个节点的行进成本。默认实现返回 new_cost。重载此方法以调整算法的行为。
- update_node(index, new_index, offset_length)#
当更新节点时,会在将 new_index 推送到堆上并更新回溯映射后立即调用此方法。
可以重载此方法,以跟踪算法的特定实现使用的其他数组。例如,MCP_Connect 类使用它来更新 ID 映射。
- class skimage.graph.MCP_Geometric(costs, offsets=None, fully_connected=True)#
基类:
MCP查找通过 n 维成本数组的距离加权最小成本路径。
有关完整详细信息,请参阅 MCP 的文档。此类与 MCP 的区别在于,路径的成本不仅仅是沿该路径的成本之和。
此类改为假定 costs 数组在每个位置都包含在该位置上行进单位距离的“成本”。例如,从 (1, 1) 到 (1, 2) 的移动(在二维中)假定起源于像素 (1, 1) 的中心,并终止于 (1, 2) 的中心。整个移动的距离为 1,一半经过 (1, 1),一半经过 (1, 2);因此,该移动的成本为
(1/2)*costs[1,1] + (1/2)*costs[1,2]。另一方面,从 (1, 1) 到 (2, 2) 的移动是沿对角线进行的,长度为 sqrt(2)。此移动的一半在像素 (1, 1) 内,另一半在 (2, 2) 内,因此此移动的成本计算为
(sqrt(2)/2)*costs[1,1] + (sqrt(2)/2)*costs[2,2]。这些计算对于大小大于 1 的偏移量没有多大意义。使用
sampling参数来处理各向异性数据。- __init__(costs, offsets=None, fully_connected=True, sampling=None)#
请参阅类文档。
- find_costs(starts, ends=None, find_all_ends=True, max_coverage=1.0, max_cumulative_cost=None, max_cost=None)#
查找从给定起始点到指定结束点的最小成本路径。
此方法查找从任何一个指定的起始索引到指定结束索引的最小成本路径。如果未给出结束位置,则会找到到 costs 数组中每个位置的最小成本路径。
- 参数:
- startsiterable
n 维起始索引列表(其中 n 是
costs数组的维度)。将找到到最近/最便宜的起始点的最小成本路径。- endsiterable, optional
n 维结束索引列表。
- find_all_endsbool, optional
如果为“True”(默认),则会找到到每个指定结束位置的最小成本路径;否则,当找到到任何结束位置的路径时,算法将停止。(如果未指定
ends,则此参数不起作用。)
- 返回:
- cumulative_costsndarray
与
costs数组的形状相同;此数组记录从最近/最便宜的起始索引到每个被考虑的索引的最小成本路径。(如果指定了ends,则并非数组中的所有元素都会被考虑:未评估的位置的累积成本将为 inf。如果find_all_ends为“False”,则只有一个指定的结束位置具有有限的累积成本。)- tracebackndarray
与
costs数组形状相同的数组;此数组包含从前驱索引到任何给定索引的偏移量。偏移量索引索引到offsets属性,该属性是一个 n 维偏移量数组。在二维情况下,如果 offsets[traceback[x, y]] 是 (-1, -1),则表示从最小成本路径到某个起始位置,[x, y] 的前驱是 [x+1, y+1]。请注意,如果 offset_index 为 -1,则表示未考虑给定的索引。
- goal_reached(index, cumcost)#
int goal_reached(int index, float cumcost) 在从堆中弹出一个索引后,在检查邻居之前,每次迭代都会调用此方法。
可以重载此方法以修改 MCP 算法的行为。一个示例可能是在达到某个累积成本或前沿距离种子点一定距离时停止算法。
如果算法不应检查当前点的邻居,则此方法应返回 1;如果算法现在已完成,则应返回 2。
- offsets#
- traceback(end)#
通过预先计算的回溯数组追踪最小成本路径。
此便捷函数从提供给 find_costs() 的起始索引之一重建到给定结束位置的最小成本路径,find_costs() 必须事先调用。在 find_costs() 运行后,可以根据需要多次调用此函数。
- 参数:
- enditerable
一个到
costs数组的 n 维索引。
- 返回:
- tracebackn 维元组列表
一个到
costs数组的索引列表,从传递给 find_costs() 的起始位置之一开始,到给定的end索引结束。这些索引指定从任何给定起始索引到end索引的最小成本路径。(该路径的总成本可以从 find_costs() 返回的cumulative_costs数组中读取。)
- class skimage.graph.RAG(label_image=None, connectivity=1, data=None, **attr)[源代码]#
基类:
Graph图像的区域邻接图 (RAG),子类
networkx.Graph。- 参数:
- label_imageint 数组
初始分割,其中每个区域标记为不同的整数。
label_image中的每个唯一值都将对应图中的一个节点。- connectivityint,取值范围为 {1, …,
label_image.ndim},可选 label_image中像素之间的连通性。对于 2D 图像,连通性为 1 对应于上下左右的直接邻居,而连通性为 2 还包括对角邻居。请参阅scipy.ndimage.generate_binary_structure()。- data
networkx.Graph规范,可选 要传递给
networkx.Graph构造函数的初始边或附加边。有效的边规范包括边列表(元组列表)、NumPy 数组和 SciPy 稀疏矩阵。- **attr关键字参数,可选
要添加到图的其他属性。
- __init__(label_image=None, connectivity=1, data=None, **attr)[源代码]#
使用边、名称或图属性初始化图。
- 参数:
- incoming_graph_data输入图(可选,默认值:None)
用于初始化图的数据。如果为 None(默认),则创建一个空图。数据可以是边列表或任何 NetworkX 图对象。如果安装了相应的可选 Python 包,则数据也可以是 2D NumPy 数组、SciPy 稀疏数组或 PyGraphviz 图。
- attr关键字参数,可选(默认值=无属性)
要作为 key=value 对添加到图的属性。
另请参阅
转换
示例
>>> G = nx.Graph() # or DiGraph, MultiGraph, MultiDiGraph, etc >>> G = nx.Graph(name="my graph") >>> e = [(1, 2), (2, 3), (3, 4)] # list of edges >>> G = nx.Graph(e)
可以分配任意图属性对 (key=value)
>>> G = nx.Graph(e, day="Friday") >>> G.graph {'day': 'Friday'}
- add_edges_from(ebunch_to_add, **attr)[源代码]#
添加 ebunch_to_add 中的所有边。
- 参数:
- ebunch_to_add边容器
容器中给出的每条边都将添加到图中。边必须以 2 元组 (u, v) 或 3 元组 (u, v, d) 的形式给出,其中 d 是包含边数据的字典。
- attr关键字参数,可选
可以使用关键字参数分配边数据(或标签或对象)。
另请参阅
add_edge添加单条边
add_weighted_edges_from添加加权边的便捷方式
注释
两次添加同一条边没有任何效果,但在每次添加重复边时都会更新任何边数据。
ebunch 中指定的边属性优先于通过关键字参数指定的属性。
从迭代器添加正在更改的图的边时,可能会引发
RuntimeError,并显示消息:RuntimeError:在迭代期间字典更改了大小。当在迭代期间修改图的底层字典时,会发生这种情况。要避免此错误,请将迭代器评估为单独的对象,例如使用list(iterator_of_edges),并将此对象传递给G.add_edges_from。示例
>>> G = nx.Graph() # or DiGraph, MultiGraph, MultiDiGraph, etc >>> G.add_edges_from([(0, 1), (1, 2)]) # using a list of edge tuples >>> e = zip(range(0, 3), range(1, 4)) >>> G.add_edges_from(e) # Add the path graph 0-1-2-3
将数据与边关联
>>> G.add_edges_from([(1, 2), (2, 3)], weight=3) >>> G.add_edges_from([(3, 4), (1, 4)], label="WN2898")
如果使用迭代器来修改同一图,则评估该图上的迭代器
>>> G = nx.Graph([(1, 2), (2, 3), (3, 4)]) >>> # Grow graph by one new node, adding edges to all existing nodes. >>> # wrong way - will raise RuntimeError >>> # G.add_edges_from(((5, n) for n in G.nodes)) >>> # correct way - note that there will be no self-edge for node 5 >>> G.add_edges_from(list((5, n) for n in G.nodes))
- add_nodes_from(nodes_for_adding, **attr)[源代码]#
添加多个节点。
- 参数:
- nodes_for_adding可迭代容器
节点容器(列表、字典、集合等)。或 (节点、属性字典) 元组的容器。使用属性字典更新节点属性。
- attr关键字参数,可选(默认值=无属性)
更新节点中所有节点的属性。在节点中指定为元组的节点属性优先于通过关键字参数指定的属性。
另请参阅
注释
从迭代器添加正在更改的图的节点时,可能会引发
RuntimeError,并显示消息:RuntimeError:在迭代期间字典更改了大小。当在迭代期间修改图的底层字典时,会发生这种情况。要避免此错误,请将迭代器评估为单独的对象,例如使用list(iterator_of_nodes),并将此对象传递给G.add_nodes_from。示例
>>> G = nx.Graph() # or DiGraph, MultiGraph, MultiDiGraph, etc >>> G.add_nodes_from("Hello") >>> K3 = nx.Graph([(0, 1), (1, 2), (2, 0)]) >>> G.add_nodes_from(K3) >>> sorted(G.nodes(), key=str) [0, 1, 2, 'H', 'e', 'l', 'o']
使用关键字更新每个节点的特定节点属性。
>>> G.add_nodes_from([1, 2], size=10) >>> G.add_nodes_from([3, 4], weight=0.4)
使用 (节点,attrdict) 元组更新特定节点的属性。
>>> G.add_nodes_from([(1, dict(size=11)), (2, {"color": "blue"})]) >>> G.nodes[1]["size"] 11 >>> H = nx.Graph() >>> H.add_nodes_from(G.nodes(data=True)) >>> H.nodes[1]["size"] 11
如果使用迭代器来修改同一图,则评估该图上的迭代器
>>> G = nx.Graph([(0, 1), (1, 2), (3, 4)]) >>> # wrong way - will raise RuntimeError >>> # G.add_nodes_from(n + 1 for n in G.nodes) >>> # correct way >>> G.add_nodes_from(list(n + 1 for n in G.nodes))
- add_weighted_edges_from(ebunch_to_add, weight='weight', **attr)[source]#
在
ebunch_to_add中添加具有指定权重属性的加权边- 参数:
- ebunch_to_add边容器
列表中或容器中给出的每个边都将被添加到图中。边必须以 3 元组 (u, v, w) 的形式给出,其中 w 是一个数字。
- weight字符串,可选(默认值= 'weight')
要添加的边权重的属性名称。
- attr关键字参数,可选(默认值=无属性)
要为所有边添加/更新的边属性。
另请参阅
add_edge添加单条边
add_edges_from添加多个边
注释
对于 Graph/DiGraph,重复添加相同的边只会更新边数据。对于 MultiGraph/MultiDiGraph,会存储重复的边。
当从迭代器添加边到你正在更改的图时,可能会引发
RuntimeError,并显示消息:RuntimeError: 在迭代期间字典大小已更改。当在迭代期间修改图的底层字典时,就会发生这种情况。为了避免此错误,请将迭代器评估为一个单独的对象,例如使用list(iterator_of_edges),并将此对象传递给G.add_weighted_edges_from。示例
>>> G = nx.Graph() # or DiGraph, MultiGraph, MultiDiGraph, etc >>> G.add_weighted_edges_from([(0, 1, 3.0), (1, 2, 7.5)])
在传递之前评估边的迭代器
>>> G = nx.Graph([(1, 2), (2, 3), (3, 4)]) >>> weight = 0.1 >>> # Grow graph by one new node, adding edges to all existing nodes. >>> # wrong way - will raise RuntimeError >>> # G.add_weighted_edges_from(((5, n, weight) for n in G.nodes)) >>> # correct way - note that there will be no self-edge for node 5 >>> G.add_weighted_edges_from(list((5, n, weight) for n in G.nodes))
- 属性 adj#
图的邻接对象,保存每个节点的邻居。
此对象是一个只读的类字典结构,具有节点键和邻居字典值。邻居字典的键是邻居,值是边数据字典。因此,
G.adj[3][2]['color'] = 'blue'将边(3, 2)的颜色设置为"blue"。迭代 G.adj 的行为类似于字典。有用的习惯用法包括
for nbr, datadict in G.adj[n].items():。邻居信息也可以通过下标访问图来提供。因此
for nbr, foovalue in G[node].data('foo', default=1):也可以工作。对于有向图,
G.adj保存传出(后继)信息。
- adjacency()[source]#
返回所有节点的(节点,邻接字典)元组的迭代器。
对于有向图,仅包含传出的邻居/邻接关系。
- 返回:
- adj_iter迭代器
图中所有节点的(节点,邻接字典)迭代器。
示例
>>> G = nx.path_graph(4) # or DiGraph, MultiGraph, MultiDiGraph, etc >>> [(n, nbrdict) for n, nbrdict in G.adjacency()] [(0, {1: {}}), (1, {0: {}, 2: {}}), (2, {1: {}, 3: {}}), (3, {2: {}})]
- clear()[source]#
从图中删除所有节点和边。
这也会删除名称以及所有图、节点和边属性。
示例
>>> G = nx.path_graph(4) # or DiGraph, MultiGraph, MultiDiGraph, etc >>> G.clear() >>> list(G.nodes) [] >>> list(G.edges) []
- clear_edges()[source]#
从图中删除所有边,而不更改节点。
示例
>>> G = nx.path_graph(4) # or DiGraph, MultiGraph, MultiDiGraph, etc >>> G.clear_edges() >>> list(G.nodes) [0, 1, 2, 3] >>> list(G.edges) []
- 属性 degree#
图的 DegreeView,表示为 G.degree 或 G.degree()。
节点度是与该节点相邻的边数。加权节点度是与该节点关联的边的边权重的总和。
此对象为(节点、度)提供了一个迭代器,以及查找单个节点的度的功能。
- 参数:
- nbunch单个节点、容器或所有节点(默认值= 所有节点)
该视图将仅报告与这些节点关联的边。
- weight字符串或 None,可选(默认值=None)
保存用作权重的数值的边属性的名称。如果为 None,则每条边的权重为 1。度是与节点相邻的边权重的总和。
- 返回:
- DegreeView 或 int
如果请求多个节点(默认值),则返回一个
DegreeView,将节点映射到它们的度。如果请求单个节点,则返回该节点的度作为整数。
示例
>>> G = nx.path_graph(4) # or DiGraph, MultiGraph, MultiDiGraph, etc >>> G.degree[0] # node 0 has degree 1 1 >>> list(G.degree([0, 1, 2])) [(0, 1), (1, 2), (2, 2)]
- edge_subgraph(edges)[source]#
返回由指定边诱导的子图。
诱导的子图包含
edges中的每个边以及与这些边中的任何一个关联的每个节点。- 参数:
- edges可迭代对象
此图中边的可迭代对象。
- 返回:
- G图
此图的边诱导子图,具有相同的边属性。
注释
返回的子图视图中的图、边和节点属性是对原始图中相应属性的引用。视图是只读的。
要创建具有自己的边或节点属性副本的子图的完整图版本,请使用
G.edge_subgraph(edges).copy()
示例
>>> G = nx.path_graph(5) >>> H = G.edge_subgraph([(0, 1), (3, 4)]) >>> list(H.nodes) [0, 1, 3, 4] >>> list(H.edges) [(0, 1), (3, 4)]
- 属性 edges#
图的 EdgeView,表示为 G.edges 或 G.edges()。
edges(self, nbunch=None, data=False, default=None)
EdgeView 对边元组提供类似集合的操作,以及边属性查找。调用时,它还提供一个 EdgeDataView 对象,该对象允许控制对边属性的访问(但不提供类似集合的操作)。因此,
G.edges[u, v]['color']提供边(u, v)的颜色属性的值,而for (u, v, c) in G.edges.data('color', default='red'):迭代所有边,如果不存在颜色属性,则生成默认值为'red'的颜色属性。- 参数:
- nbunch单个节点、容器或所有节点(默认值= 所有节点)
该视图将仅报告来自这些节点的边。
- data字符串或布尔值,可选(默认值=False)
在 3 元组 (u, v, ddict[data]) 中返回的边属性。如果为 True,则在 3 元组 (u, v, ddict) 中返回边属性字典。如果为 False,则返回 2 元组 (u, v)。
- default值,可选(默认值=None)
用于没有请求属性的边的值。仅当 data 不为 True 或 False 时才相关。
- 返回:
- edgesEdgeView
边属性的视图,通常迭代边的 (u, v) 或 (u, v, d) 元组,但也可以用于属性查找,如
edges[u, v]['foo']。
注释
nbunch 中不在图中的节点将被(静默地)忽略。对于有向图,这将返回出边。
示例
>>> G = nx.path_graph(3) # or MultiGraph, etc >>> G.add_edge(2, 3, weight=5) >>> [e for e in G.edges] [(0, 1), (1, 2), (2, 3)] >>> G.edges.data() # default data is {} (empty dict) EdgeDataView([(0, 1, {}), (1, 2, {}), (2, 3, {'weight': 5})]) >>> G.edges.data("weight", default=1) EdgeDataView([(0, 1, 1), (1, 2, 1), (2, 3, 5)]) >>> G.edges([0, 3]) # only edges from these nodes EdgeDataView([(0, 1), (3, 2)]) >>> G.edges(0) # only edges from node 0 EdgeDataView([(0, 1)])
- fresh_copy()[source]#
返回具有相同数据结构的全新副本图。
全新副本没有节点、边或图属性。它与当前图具有相同的数据结构。此方法通常用于创建图的空版本。
在用 networkx v2 对 Graph 进行子类化时需要这样做,并且不会为 v1 带来问题。以下是从 network 1.x 迁移到 2.x 文档中的更多详细信息
With the new GraphViews (SubGraph, ReversedGraph, etc) you can't assume that ``G.__class__()`` will create a new instance of the same graph type as ``G``. In fact, the call signature for ``__class__`` differs depending on whether ``G`` is a view or a base class. For v2.x you should use ``G.fresh_copy()`` to create a null graph of the correct type---ready to fill with nodes and edges.
- get_edge_data(u, v, default=None)[source]#
返回与边 (u, v) 关联的属性字典。
这与
G[u][v]相同,但如果该边不存在,则返回默认值而不是异常。- 参数:
- u, v节点
- default:任何 Python 对象(默认值=None)
如果未找到边 (u, v),则返回该值。
- 返回:
- edge_dict字典
边属性字典。
示例
>>> G = nx.path_graph(4) # or DiGraph, MultiGraph, MultiDiGraph, etc >>> G[0][1] {}
警告:不允许赋值给
G[u][v]。但是,可以安全地赋值属性G[u][v]['foo']>>> G[0][1]["weight"] = 7 >>> G[0][1]["weight"] 7 >>> G[1][0]["weight"] 7
>>> G = nx.path_graph(4) # or DiGraph, MultiGraph, MultiDiGraph, etc >>> G.get_edge_data(0, 1) # default edge data is {} {} >>> e = (0, 1) >>> G.get_edge_data(*e) # tuple form {} >>> G.get_edge_data("a", "b", default=0) # edge not in graph, return 0 0
- has_edge(u, v)[源代码]#
如果边 (u, v) 在图中,则返回 True。
这与
v in G[u]相同,但没有 KeyError 异常。- 参数:
- u, v节点
节点可以是字符串或数字等。节点必须是可哈希的 (且不是 None) Python 对象。
- 返回:
- edge_ind布尔值
如果边在图中,则为 True,否则为 False。
示例
>>> G = nx.path_graph(4) # or DiGraph, MultiGraph, MultiDiGraph, etc >>> G.has_edge(0, 1) # using two nodes True >>> e = (0, 1) >>> G.has_edge(*e) # e is a 2-tuple (u, v) True >>> e = (0, 1, {"weight": 7}) >>> G.has_edge(*e[:2]) # e is a 3-tuple (u, v, data_dictionary) True
以下语法等效
>>> G.has_edge(0, 1) True >>> 1 in G[0] # though this gives KeyError if 0 not in G True
- has_node(n)[源代码]#
如果图包含节点 n,则返回 True。
与
n in G相同- 参数:
- n节点
示例
>>> G = nx.path_graph(3) # or DiGraph, MultiGraph, MultiDiGraph, etc >>> G.has_node(0) True
使用以下方法更易读且更简单
>>> 0 in G True
- merge_nodes(src, dst, weight_func=<function min_weight>, in_place=True, extra_arguments=None, extra_keywords=None)[源代码]#
合并节点
src和dst。新的组合节点与
src和dst的所有邻居相邻。调用weight_func来确定新节点上的边的权重。- 参数:
- src, dst整数
要合并的节点。
- weight_func可调用对象,可选
用于确定与新节点关联的边的属性的函数。对于
src和dst的每个邻居n,将按如下方式调用weight_func:weight_func(src, dst, n, *extra_arguments, **extra_keywords)。src、dst和n是 RAG 对象中顶点的 ID,而 RAG 对象又是networkx.Graph的子类。它应该返回结果边的属性字典。- in_placebool,可选
如果设置为
True,则合并后的节点具有 iddst,否则合并后的节点具有新的 id,该 id 将被返回。- extra_arguments序列,可选
传递给
weight_func的额外位置参数的序列。- extra_keywords字典,可选
传递给
weight_func的关键字参数的字典。
- 返回:
- id整数
新节点的 id。
注释
如果
in_place为False,则生成的节点具有新的 id,而不是dst。
- property name#
图的字符串标识符。
此图属性出现在由字符串
"name"键控的属性字典 G.graph 中,以及属性(技术上是属性)G.name中。这完全由用户控制。
- nbunch_iter(nbunch=None)[源代码]#
返回一个迭代器,该迭代器遍历 nbunch 中也位于图中的节点。
将检查 nbunch 中的节点是否属于该图,如果不是,则会静默忽略。
- 参数:
- nbunch单个节点、容器或所有节点(默认值= 所有节点)
该视图将仅报告与这些节点关联的边。
- 返回:
- niter迭代器
一个迭代器,它遍历 nbunch 中也位于图中的节点。如果 nbunch 为 None,则遍历图中的所有节点。
- 引发:
- NetworkXError
如果 nbunch 不是节点或节点序列。如果 nbunch 中的节点不可哈希。
另请参阅
Graph.__iter__
注释
当 nbunch 是迭代器时,返回的迭代器直接从 nbunch 产生值,并在 nbunch 耗尽时变为耗尽。
要测试 nbunch 是否是单个节点,即使在使用此例程处理后,也可以使用“if nbunch in self:”。
如果 nbunch 不是节点或(可能为空的)序列/迭代器或 None,则会引发
NetworkXError。此外,如果 nbunch 中的任何对象不可哈希,也会引发NetworkXError。
- neighbors(n)[源代码]#
返回节点 n 的所有邻居的迭代器。
这与
iter(G[n])相同- 参数:
- n节点
图中的一个节点
- 返回:
- neighbors迭代器
节点 n 的所有邻居的迭代器
- 引发:
- NetworkXError
如果节点 n 不在图中。
注释
访问邻居的替代方法是
G.adj[n]或G[n]>>> G = nx.Graph() # or DiGraph, MultiGraph, MultiDiGraph, etc >>> G.add_edge("a", "b", weight=7) >>> G["a"] AtlasView({'b': {'weight': 7}}) >>> G = nx.path_graph(4) >>> [n for n in G[0]] [1]
示例
>>> G = nx.path_graph(4) # or DiGraph, MultiGraph, MultiDiGraph, etc >>> [n for n in G.neighbors(0)] [1]
- property nodes#
作为 G.nodes 或 G.nodes() 的图的 NodeView。
可以用作
G.nodes用于数据查找和类似集合的操作。也可以用作G.nodes(data='color', default=None),以返回 NodeDataView,该视图报告特定的节点数据,但不进行集合操作。它还提供了一个类似字典的接口,G.nodes.items()迭代(node, nodedata)二元组,而G.nodes[3]['foo']提供节点3的foo属性的值。此外,视图G.nodes.data('foo')提供每个节点的foo属性的类似字典的接口。G.nodes.data('foo', default=1)为不具有属性foo的节点提供默认值。- 参数:
- data字符串或布尔值,可选(默认值=False)
在 2 元组 (n, ddict[data]) 中返回的节点属性。如果为 True,则返回整个节点属性字典作为 (n, ddict)。如果为 False,则仅返回节点 n。
- default值,可选(默认值=None)
用于不具有请求属性的节点的值。仅在 data 不是 True 或 False 时相关。
- 返回:
- NodeView
允许对节点进行类似集合的操作,以及节点属性字典查找和调用以获取 NodeDataView。NodeDataView 迭代
(n, data),并且没有集合操作。NodeView 迭代n,并且包含集合操作。调用时,如果 data 为 False,则返回一个节点迭代器。否则返回 2 元组 (节点, 属性值) 的迭代器,其中属性在
data中指定。如果 data 为 True,则属性变为整个数据字典。
注释
如果不需要节点数据,则使用表达式
for n in G或list(G)更简单且等效。示例
有两种简单的方法可以获取图中所有节点的列表
>>> G = nx.path_graph(3) >>> list(G.nodes) [0, 1, 2] >>> list(G) [0, 1, 2]
要获取节点数据以及节点
>>> G.add_node(1, time="5pm") >>> G.nodes[0]["foo"] = "bar" >>> list(G.nodes(data=True)) [(0, {'foo': 'bar'}), (1, {'time': '5pm'}), (2, {})] >>> list(G.nodes.data()) [(0, {'foo': 'bar'}), (1, {'time': '5pm'}), (2, {})]
>>> list(G.nodes(data="foo")) [(0, 'bar'), (1, None), (2, None)] >>> list(G.nodes.data("foo")) [(0, 'bar'), (1, None), (2, None)]
>>> list(G.nodes(data="time")) [(0, None), (1, '5pm'), (2, None)] >>> list(G.nodes.data("time")) [(0, None), (1, '5pm'), (2, None)]
>>> list(G.nodes(data="time", default="Not Available")) [(0, 'Not Available'), (1, '5pm'), (2, 'Not Available')] >>> list(G.nodes.data("time", default="Not Available")) [(0, 'Not Available'), (1, '5pm'), (2, 'Not Available')]
如果某些节点具有属性,而其余节点假定具有默认属性值,则可以使用
default关键字参数从节点/属性对创建字典,以保证该值永远不会为 None>>> G = nx.Graph() >>> G.add_node(0) >>> G.add_node(1, weight=2) >>> G.add_node(2, weight=3) >>> dict(G.nodes(data="weight", default=1)) {0: 1, 1: 2, 2: 3}
- number_of_edges(u=None, v=None)[source]#
返回两个节点之间的边数。
- 参数:
- u, v节点,可选 (默认=所有边)
如果指定了 u 和 v,则返回 u 和 v 之间的边数。否则返回所有边的总数。
- 返回:
- nedgesint
图中的边数。如果指定了节点
u和v,则返回这些节点之间的边数。如果图是有向的,则仅返回从u到v的边数。
另请参阅
示例
对于无向图,此方法计算图中边的总数
>>> G = nx.path_graph(4) >>> G.number_of_edges() 3
如果指定了两个节点,则此方法计算连接这两个节点的边的总数
>>> G.number_of_edges(0, 1) 1
对于有向图,此方法可以计算从
u到v的有向边的总数>>> G = nx.DiGraph() >>> G.add_edge(0, 1) >>> G.add_edge(1, 0) >>> G.number_of_edges(0, 1) 1
- number_of_nodes()[source]#
返回图中的节点数。
- 返回:
- nnodesint
图中的节点数。
另请参阅
阶数相同的方法
__len__相同的方法
示例
>>> G = nx.path_graph(3) # or DiGraph, MultiGraph, MultiDiGraph, etc >>> G.number_of_nodes() 3
- order()[source]#
返回图中的节点数。
- 返回:
- nnodesint
图中的节点数。
另请参阅
number_of_nodes相同的方法
__len__相同的方法
示例
>>> G = nx.path_graph(3) # or DiGraph, MultiGraph, MultiDiGraph, etc >>> G.order() 3
- remove_edge(u, v)[source]#
删除 u 和 v 之间的边。
- 参数:
- u, v节点
删除节点 u 和 v 之间的边。
- 引发:
- NetworkXError
如果 u 和 v 之间没有边。
另请参阅
remove_edges_from删除一组边
示例
>>> G = nx.path_graph(4) # or DiGraph, etc >>> G.remove_edge(0, 1) >>> e = (1, 2) >>> G.remove_edge(*e) # unpacks e from an edge tuple >>> e = (2, 3, {"weight": 7}) # an edge with attribute data >>> G.remove_edge(*e[:2]) # select first part of edge tuple
- remove_edges_from(ebunch)[source]#
删除 ebunch 中指定的所有边。
- 参数:
- ebunch:边元组的列表或容器
列表中或容器中给出的每条边都将从图中删除。边可以是
2 元组 (u, v) 表示 u 和 v 之间的边。
3 元组 (u, v, k),其中 k 被忽略。
另请参阅
remove_edge删除单条边
注释
如果 ebunch 中的边不在图中,则会静默失败。
示例
>>> G = nx.path_graph(4) # or DiGraph, MultiGraph, MultiDiGraph, etc >>> ebunch = [(1, 2), (2, 3)] >>> G.remove_edges_from(ebunch)
- remove_node(n)[source]#
删除节点 n。
删除节点 n 以及所有相邻的边。尝试删除不存在的节点将引发异常。
- 参数:
- n节点
图中的一个节点
- 引发:
- NetworkXError
如果 n 不在图中。
另请参阅
示例
>>> G = nx.path_graph(3) # or DiGraph, MultiGraph, MultiDiGraph, etc >>> list(G.edges) [(0, 1), (1, 2)] >>> G.remove_node(1) >>> list(G.edges) []
- remove_nodes_from(nodes)[source]#
删除多个节点。
- 参数:
- nodes可迭代容器
节点容器(列表、字典、集合等)。如果容器中的节点不在图中,则会静默忽略。
另请参阅
注释
从正在更改的图的迭代器中删除节点时,会引发带有消息的
RuntimeError:RuntimeError: dictionary changed size during iteration。当在迭代期间修改图的基础字典时,会发生这种情况。要避免此错误,请将迭代器评估为单独的对象,例如通过使用list(iterator_of_nodes),并将此对象传递给G.remove_nodes_from。示例
>>> G = nx.path_graph(3) # or DiGraph, MultiGraph, MultiDiGraph, etc >>> e = list(G.nodes) >>> e [0, 1, 2] >>> G.remove_nodes_from(e) >>> list(G.nodes) []
如果使用迭代器来修改同一图,则评估该图上的迭代器
>>> G = nx.Graph([(0, 1), (1, 2), (3, 4)]) >>> # this command will fail, as the graph's dict is modified during iteration >>> # G.remove_nodes_from(n for n in G.nodes if n < 2) >>> # this command will work, since the dictionary underlying graph is not modified >>> G.remove_nodes_from(list(n for n in G.nodes if n < 2))
- size(weight=None)[source]#
返回边数或所有边权重的总和。
- 参数:
- weight字符串或 None,可选(默认值=None)
边属性,其中包含用作权重的数值。如果为 None,则每条边的权重为 1。
- 返回:
- 大小数值
边数或(如果提供了 weight 关键字)总权重之和。
如果 weight 为 None,则返回 int。否则返回 float(或者如果权重更通用,则返回更通用的数值)。
另请参阅
示例
>>> G = nx.path_graph(4) # or DiGraph, MultiGraph, MultiDiGraph, etc >>> G.size() 3
>>> G = nx.Graph() # or DiGraph, MultiGraph, MultiDiGraph, etc >>> G.add_edge("a", "b", weight=2) >>> G.add_edge("b", "c", weight=4) >>> G.size() 2 >>> G.size(weight="weight") 6.0
- subgraph(nodes)[source]#
返回由
nodes诱导的子图的 SubGraph 视图。图的诱导子图包含
nodes中的节点以及这些节点之间的边。- 参数:
- nodes列表,可迭代对象
将遍历一次的节点容器。
- 返回:
- GSubGraph 视图
图的子图视图。不能更改图结构,但是可以更改节点/边属性,并且它们与原始图共享。
注释
图、边和节点属性与原始图共享。视图排除了对图结构的更改,但是对属性的更改会反映在原始图中。
要创建具有边/节点属性的自身副本的子图,请使用:G.subgraph(nodes).copy()
要将图就地缩小为子图,可以删除节点:G.remove_nodes_from([n for n in G if n not in set(nodes)])
子图视图有时不是你想要的。在大多数情况下,当你想要做的不仅仅是简单地查看诱导的边时,将子图创建为自己的图更有意义,代码如下所示
# Create a subgraph SG based on a (possibly multigraph) G SG = G.__class__() SG.add_nodes_from((n, G.nodes[n]) for n in largest_wcc) if SG.is_multigraph(): SG.add_edges_from( (n, nbr, key, d) for n, nbrs in G.adj.items() if n in largest_wcc for nbr, keydict in nbrs.items() if nbr in largest_wcc for key, d in keydict.items() ) else: SG.add_edges_from( (n, nbr, d) for n, nbrs in G.adj.items() if n in largest_wcc for nbr, d in nbrs.items() if nbr in largest_wcc ) SG.graph.update(G.graph)
示例
>>> G = nx.path_graph(4) # or DiGraph, MultiGraph, MultiDiGraph, etc >>> H = G.subgraph([0, 1, 2]) >>> list(H.edges) [(0, 1), (1, 2)]
- to_directed(as_view=False)[source]#
返回图的有向表示。
- 返回:
- GDiGraph
一个有向图,具有相同的名称,相同的节点,并且每条边 (u, v, data) 都由两条有向边 (u, v, data) 和 (v, u, data) 替换。
注释
这会返回边、节点和图属性的“深层副本”,该副本会尝试完全复制所有数据和引用。
这与类似的 D=DiGraph(G) 形成对比,后者返回数据的浅层副本。
有关浅拷贝和深拷贝的更多信息,请参阅 Python copy 模块:https://docs.pythonlang.cn/3/library/copy.html。
警告:如果已将 Graph 子类化以在数据结构中使用类似字典的对象,则这些更改不会传递到此方法创建的 DiGraph 中。
示例
>>> G = nx.Graph() # or MultiGraph, etc >>> G.add_edge(0, 1) >>> H = G.to_directed() >>> list(H.edges) [(0, 1), (1, 0)]
如果已经是定向图,则返回一个(深)拷贝
>>> G = nx.DiGraph() # or MultiDiGraph, etc >>> G.add_edge(0, 1) >>> H = G.to_directed() >>> list(H.edges) [(0, 1)]
- to_directed_class()[源代码]#
返回用于创建空有向副本的类。
如果对基类进行了子类化,请使用此方法来指定用于
to_directed()副本的有向类。
- to_undirected(as_view=False)[源代码]#
返回图的无向副本。
- 参数:
- as_viewbool (可选,默认=False)
如果为 True,则返回原始无向图的视图。
- 返回:
- GGraph/MultiGraph
图的深拷贝。
另请参阅
Graph,copy,add_edge,add_edges_from
注释
这会返回边、节点和图属性的“深层副本”,该副本会尝试完全复制所有数据和引用。
这与类似的
G = nx.DiGraph(D)形成对比,后者返回数据的浅拷贝。有关浅拷贝和深拷贝的更多信息,请参阅 Python copy 模块:https://docs.pythonlang.cn/3/library/copy.html。
警告:如果已将 DiGraph 子类化以在数据结构中使用类似字典的对象,则这些更改不会传递到此方法创建的 Graph 中。
示例
>>> G = nx.path_graph(2) # or MultiGraph, etc >>> H = G.to_directed() >>> list(H.edges) [(0, 1), (1, 0)] >>> G2 = H.to_undirected() >>> list(G2.edges) [(0, 1)]
- to_undirected_class()[源代码]#
返回用于创建空无向副本的类。
如果对基类进行了子类化,请使用此方法来指定用于
to_directed()副本的有向类。
- update(edges=None, nodes=None)[源代码]#
使用节点/边/图作为输入来更新图。
与 dict.update 类似,此方法将图作为输入,将图的节点和边添加到此图中。它还可以接受两个输入:边和节点。最后,它可以接受边或节点。要仅指定节点,必须使用关键字
nodes。边和节点的集合的处理方式类似于 add_edges_from/add_nodes_from 方法。在迭代时,它们应该产生 2 元组 (u, v) 或 3 元组 (u, v, datadict)。
- 参数:
另请参阅
add_edges_from将多条边添加到图中
add_nodes_from将多个节点添加到图中
注释
如果要使用邻接结构更新图,则可以很容易地从邻接结构中获取边/节点。以下示例提供了常见情况,您的邻接结构可能略有不同,需要对这些示例进行调整
>>> # dict-of-set/list/tuple >>> adj = {1: {2, 3}, 2: {1, 3}, 3: {1, 2}} >>> e = [(u, v) for u, nbrs in adj.items() for v in nbrs] >>> G.update(edges=e, nodes=adj)
>>> DG = nx.DiGraph() >>> # dict-of-dict-of-attribute >>> adj = {1: {2: 1.3, 3: 0.7}, 2: {1: 1.4}, 3: {1: 0.7}} >>> e = [ ... (u, v, {"weight": d}) ... for u, nbrs in adj.items() ... for v, d in nbrs.items() ... ] >>> DG.update(edges=e, nodes=adj)
>>> # dict-of-dict-of-dict >>> adj = {1: {2: {"weight": 1.3}, 3: {"color": 0.7, "weight": 1.2}}} >>> e = [ ... (u, v, {"weight": d}) ... for u, nbrs in adj.items() ... for v, d in nbrs.items() ... ] >>> DG.update(edges=e, nodes=adj)
>>> # predecessor adjacency (dict-of-set) >>> pred = {1: {2, 3}, 2: {3}, 3: {3}} >>> e = [(v, u) for u, nbrs in pred.items() for v in nbrs]
>>> # MultiGraph dict-of-dict-of-dict-of-attribute >>> MDG = nx.MultiDiGraph() >>> adj = { ... 1: {2: {0: {"weight": 1.3}, 1: {"weight": 1.2}}}, ... 3: {2: {0: {"weight": 0.7}}}, ... } >>> e = [ ... (u, v, ekey, d) ... for u, nbrs in adj.items() ... for v, keydict in nbrs.items() ... for ekey, d in keydict.items() ... ] >>> MDG.update(edges=e)
示例
>>> G = nx.path_graph(5) >>> G.update(nx.complete_graph(range(4, 10))) >>> from itertools import combinations >>> edges = ( ... (u, v, {"power": u * v}) ... for u, v in combinations(range(10, 20), 2) ... if u * v < 225 ... ) >>> nodes = [1000] # for singleton, use a container >>> G.update(edges, nodes)