注意
转到末尾下载完整的示例代码。或通过 Binder 在浏览器中运行此示例
与 3D 图像(肾脏组织)交互#
在本教程中,我们将交互式探索具有三个空间维度和三个颜色维度(通道)的生物医学图像。有关 3D 图像处理的通用介绍,请参阅探索 3D 图像(细胞)。我们在此处使用的数据对应于使用共聚焦荧光显微镜成像的肾脏组织(更多详细信息请参见[1]下的kidney-tissue-fluorescence.tif)。
import matplotlib.pyplot as plt
import numpy as np
import plotly
import plotly.express as px
from skimage import data
加载图像#
此生物医学图像可通过scikit-image的数据注册表获得。
data = data.kidney()
返回的数据集是 3D 多通道图像
print(f'number of dimensions: {data.ndim}')
print(f'shape: {data.shape}')
print(f'dtype: {data.dtype}')
number of dimensions: 4
shape: (16, 512, 512, 3)
dtype: uint16
尺寸按以下顺序提供:(z, y, x, c),即[平面, 行, 列, 通道]。
n_plane, n_row, n_col, n_chan = data.shape
现在我们只考虑数据的切片(2D 平面)。更具体地说,我们考虑堆栈中间的切片。imshow函数可以显示灰度和 RGB(A) 2D 图像。
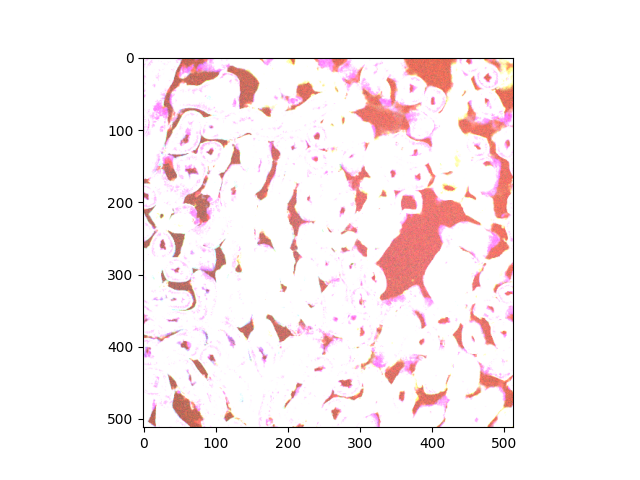
Clipping input data to the valid range for imshow with RGB data ([0..1] for floats or [0..255] for integers). Got range [64..4095].
<matplotlib.image.AxesImage object at 0x7fb7a0a61e50>
根据警告消息,值的范围是意外的。图像渲染在颜色方面显然不令人满意。
range: (10, 4095)
我们转向 Plotly 的 plotly.express.imshow() 函数的实现,因为它支持超出浮点数的 (0.0, 1.0) 和整数的 (0, 255) 的值范围。
这是 荧光显微镜图像!
规范化每个通道的范围#
一般来说,我们可能希望在每个通道的基础上规范化值的范围。让我们沿着通道轴对我们的数据(切片)进行分面。这样,我们得到三个单通道图像,其中使用每个图像的最大值
每个颜色通道的值范围是多少?我们通过在所有非通道轴上取最小值和最大值来检查。
range for channel 0: (10, 4095)
range for channel 1: (68, 4095)
range for channel 2: (35, 4095)
让我们非常具体地按通道传递值范围
Plotly 允许您通过平移、放大和缩小以及以 PNG 格式导出所需的图形来与此可视化进行交互。
将切片作为动画帧浏览#
单击播放按钮,沿着 z 轴移动,遍历所有 16 个切片的堆栈。
结合通道分面和切片动画#
fig = px.imshow(
data,
animation_frame=0,
facet_col=3,
binary_string=True,
labels={'facet_col': 'channel', 'animation_frame': 'plane'},
)
plotly.io.show(fig)
生物学家的眼睛可以看出,两个明亮的斑点(在通道=2 中最清楚地看到)是肾脏肾小球[2]。
plt.show()
脚本的总运行时间: (0 分钟 14.496 秒)
