注意
转到末尾下载完整的示例代码。或通过 Binder 在浏览器中运行此示例
使用滚动球算法估计背景强度#
滚动球算法用于估计灰度图像在曝光不均匀情况下的背景强度。它常用于生物医学图像处理,最早由 Stanley R. Sternberg 在 1983 年提出 [1]。
该算法作为滤波器工作,并且非常直观。我们将图像视为一个表面,其中每个像素的位置都堆叠了单位大小的块。块的数量,因此表面高度,由像素的强度决定。为了获得所需(像素)位置的背景强度,我们想象将一个球体浸入所需位置的表面下方。一旦它被块完全覆盖,球体的顶点就决定了该位置的背景强度。然后,我们可以在表面下方滚动这个球体,以获得整个图像的背景值。
Scikit-image 实现了此滚动球算法的通用版本,它不仅允许您使用球体,还允许使用任意形状作为内核,并且适用于 n 维 ndimage。这使您可以直接过滤 RGB 图像或沿任何(或所有)空间维度过滤图像堆栈。
经典滚动球#
在 scikit-image 中,滚动球算法假设您的背景具有低强度(黑色),而特征具有高强度(白色)。如果您的图像属于这种情况,您可以直接使用如下过滤器
import matplotlib.pyplot as plt
import numpy as np
import pywt
from skimage import data, restoration, util
def plot_result(image, background):
fig, ax = plt.subplots(nrows=1, ncols=3)
ax[0].imshow(image, cmap='gray')
ax[0].set_title('Original image')
ax[0].axis('off')
ax[1].imshow(background, cmap='gray')
ax[1].set_title('Background')
ax[1].axis('off')
ax[2].imshow(image - background, cmap='gray')
ax[2].set_title('Result')
ax[2].axis('off')
fig.tight_layout()
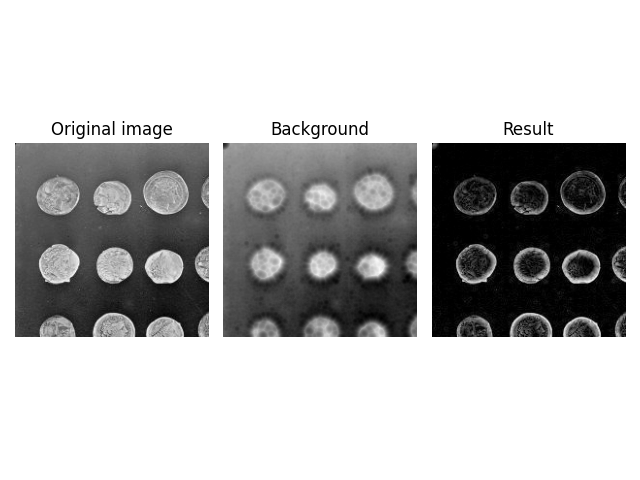
image = data.coins()
background = restoration.rolling_ball(image)
plot_result(image, background)
plt.show()

白色背景#
如果您的亮背景上有暗特征,则需要在将图像传递到算法之前反转图像,然后反转结果。这可以通过以下方式完成
image = data.page()
image_inverted = util.invert(image)
background_inverted = restoration.rolling_ball(image_inverted, radius=45)
filtered_image_inverted = image_inverted - background_inverted
filtered_image = util.invert(filtered_image_inverted)
background = util.invert(background_inverted)
fig, ax = plt.subplots(nrows=1, ncols=3)
ax[0].imshow(image, cmap='gray')
ax[0].set_title('Original image')
ax[0].axis('off')
ax[1].imshow(background, cmap='gray')
ax[1].set_title('Background')
ax[1].axis('off')
ax[2].imshow(filtered_image, cmap='gray')
ax[2].set_title('Result')
ax[2].axis('off')
fig.tight_layout()
plt.show()

小心不要在减去明亮的背景时成为整数下溢的受害者。例如,此代码看起来正确,但可能由于下溢而导致不需要的伪影。您可以在可视化的右上角看到这一点。
image = data.page()
image_inverted = util.invert(image)
background_inverted = restoration.rolling_ball(image_inverted, radius=45)
background = util.invert(background_inverted)
underflow_image = image - background # integer underflow occurs here
# correct subtraction
correct_image = util.invert(image_inverted - background_inverted)
fig, ax = plt.subplots(nrows=1, ncols=2)
ax[0].imshow(underflow_image, cmap='gray')
ax[0].set_title('Background Removal with Underflow')
ax[0].axis('off')
ax[1].imshow(correct_image, cmap='gray')
ax[1].set_title('Correct Background Removal')
ax[1].axis('off')
fig.tight_layout()
plt.show()

图像数据类型#
rolling_ball 可以处理 np.uint8 以外的数据类型。您可以通过相同的方式将它们传递给函数。
image = data.coins()[:200, :200].astype(np.uint16)
background = restoration.rolling_ball(image, radius=70.5)
plot_result(image, background)
plt.show()

但是,如果您使用已归一化为 [0, 1] 的浮点图像,则需要小心。在这种情况下,球将比图像强度大得多,这可能会导致意外的结果。
image = util.img_as_float(data.coins()[:200, :200])
background = restoration.rolling_ball(image, radius=70.5)
plot_result(image, background)
plt.show()
由于 radius=70.5 远大于图像的最大强度,因此有效内核大小会显着减小,即,只有球体的一小部分(大约 radius=10)在图像中滚动。您可以在下面的 高级形状 部分中找到对此奇怪效果的重现。
要获得预期结果,您需要降低内核的强度。这是通过使用 kernel 参数手动指定内核来完成的。
注意:半径等于椭圆的半轴长度,它是完整轴的一半。因此,内核形状乘以二。
normalized_radius = 70.5 / 255
image = util.img_as_float(data.coins())
kernel = restoration.ellipsoid_kernel((70.5 * 2, 70.5 * 2), normalized_radius * 2)
background = restoration.rolling_ball(image, kernel=kernel)
plot_result(image, background)
plt.show()

高级形状#
默认情况下,rolling_ball 使用球形内核(无需惊讶)。 有时,这可能过于局限 —— 如上面的例子所示 —— 因为强度维度与空间维度相比具有不同的尺度,或者因为图像维度可能具有不同的含义 —— 一个维度可能是图像堆栈中的堆栈计数器。
为了解决这个问题,rolling_ball 有一个 kernel 参数,允许您指定要使用的内核。内核必须具有与图像相同的维度(注意:是维度,而不是形状)。为了帮助创建内核,skimage 提供了两个默认内核。ball_kernel 指定球形内核,并用作默认内核。ellipsoid_kernel 指定椭球形内核。
image = data.coins()
kernel = restoration.ellipsoid_kernel((70.5 * 2, 70.5 * 2), 70.5 * 2)
background = restoration.rolling_ball(image, kernel=kernel)
plot_result(image, background)
plt.show()

您还可以使用 ellipsoid_kernel 来重现之前意外的结果,并看到有效的(空间)滤波器尺寸减小了。
image = data.coins()
kernel = restoration.ellipsoid_kernel((10 * 2, 10 * 2), 255 * 2)
background = restoration.rolling_ball(image, kernel=kernel)
plot_result(image, background)
plt.show()

更高维度#
rolling_ball 的另一个特点是您可以直接将其应用于更高维度的图像,例如,在共聚焦显微镜中获得的图像 z 堆栈。内核维度的数量必须与图像维度匹配,因此内核形状现在是 3 维的。
image = data.cells3d()[:, 1, ...]
background = restoration.rolling_ball(
image, kernel=restoration.ellipsoid_kernel((1, 21, 21), 0.1)
)
plot_result(image[30, ...], background[30, ...])
plt.show()
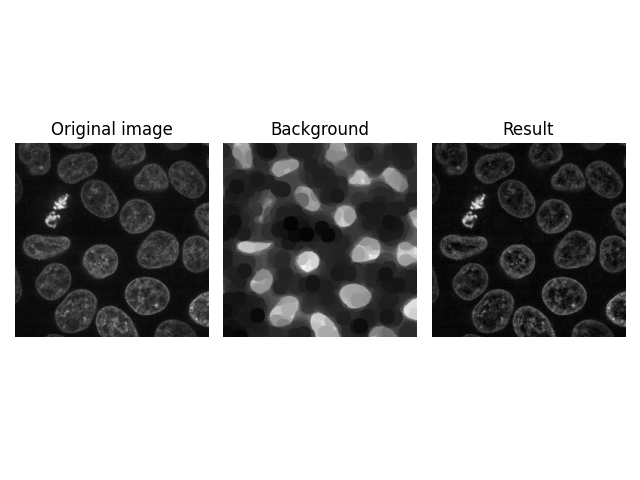
内核大小为 1 时,不会沿该轴进行滤波。换句话说,上面的滤波器单独应用于堆栈中的每个图像。
但是,您也可以通过指定一个非 1 的值来同时沿所有 3 个维度进行滤波。
image = data.cells3d()[:, 1, ...]
background = restoration.rolling_ball(
image, kernel=restoration.ellipsoid_kernel((5, 21, 21), 0.1)
)
plot_result(image[30, ...], background[30, ...])
plt.show()
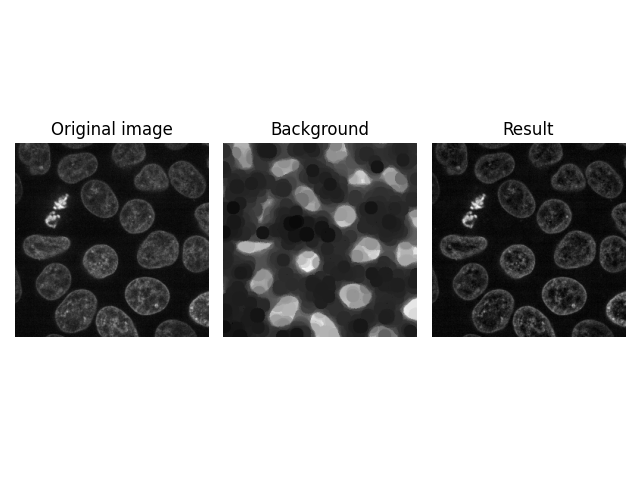
另一种可能性是沿平面轴(z 堆栈轴)滤波单个像素。
image = data.cells3d()[:, 1, ...]
background = restoration.rolling_ball(
image, kernel=restoration.ellipsoid_kernel((100, 1, 1), 0.1)
)
plot_result(image[30, ...], background[30, ...])
plt.show()
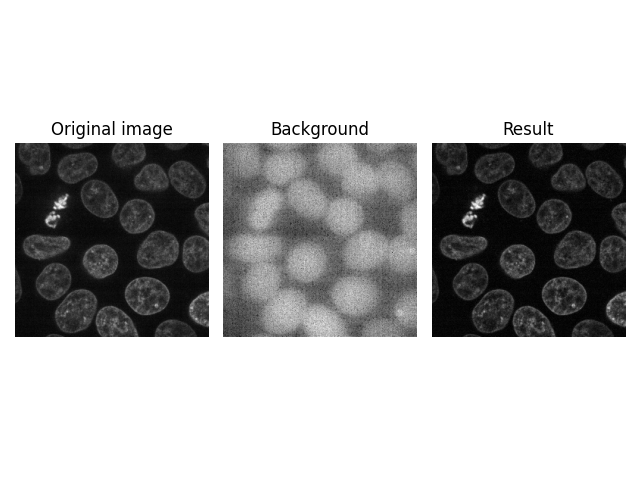
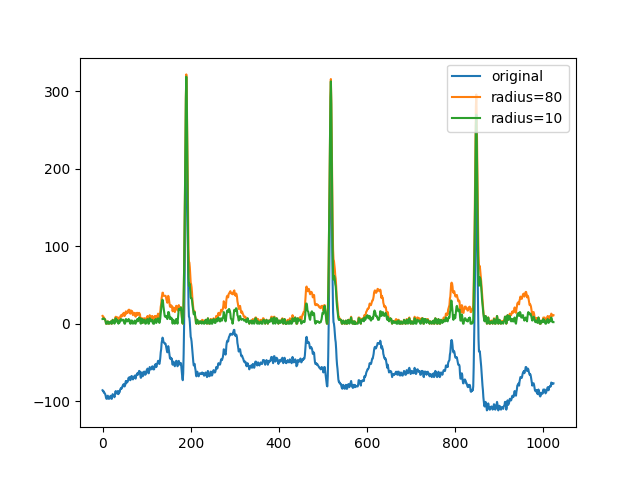
1D 信号滤波#
作为 rolling_ball 的 n 维特性的另一个示例,我们展示了 1D 数据的实现。在这里,我们有兴趣去除 ECG 波形的背景信号,以检测突出的峰值(高于局部基线的值)。可以使用较小的半径值去除较平滑的峰值。
x = pywt.data.ecg()
background = restoration.rolling_ball(x, radius=80)
background2 = restoration.rolling_ball(x, radius=10)
plt.figure()
plt.plot(x, label='original')
plt.plot(x - background, label='radius=80')
plt.plot(x - background2, label='radius=10')
plt.legend()
plt.show()
脚本的总运行时间: (0 分 43.024 秒)
