注意
转到末尾以下载完整的示例代码。或者通过 Binder 在您的浏览器中运行此示例
分割人类细胞(有丝分裂中)#
在此示例中,我们分析人类细胞的显微镜图像。我们使用 Jason Moffat [1] 通过 CellProfiler 提供的数据。
import matplotlib.pyplot as plt
import numpy as np
from scipy import ndimage as ndi
import skimage as ski
image = ski.data.human_mitosis()
fig, ax = plt.subplots()
ax.imshow(image, cmap='gray')
ax.set_title('Microscopy image of human cells stained for nuclear DNA')
plt.show()

我们可以看到黑暗背景上的许多细胞核。它们中的大多数是光滑的并且具有椭圆形。但是,我们可以区分一些较亮的点,这些点对应于正在进行有丝分裂(细胞分裂)的细胞核。
可视化灰度图像的另一种方法是轮廓图
fig, ax = plt.subplots(figsize=(5, 5))
qcs = ax.contour(image, origin='image')
ax.set_title('Contour plot of the same raw image')
plt.show()
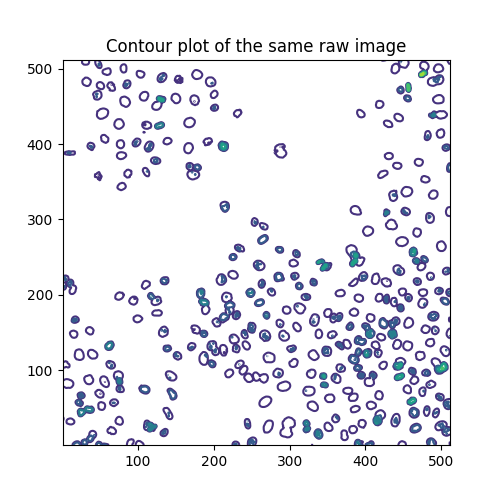
轮廓线在这些级别绘制
array([ 0., 40., 80., 120., 160., 200., 240., 280.])
每个级别分别具有以下数量的线段
# Levels without segments still contain one empty array, account for that
[len(seg) if seg[0].size else 0 for seg in qcs.allsegs]
[0, 320, 270, 48, 19, 3, 1, 0]
估计有丝分裂指数#
细胞生物学使用有丝分裂指数来量化细胞分裂,从而量化细胞增殖。根据定义,它是处于有丝分裂中的细胞数与细胞总数的比率。要分析上面的图像,我们因此对两个阈值感兴趣:一个将细胞核与背景分开,另一个将分裂细胞核(较亮的点)与未分裂细胞核分开。为了分离这三种不同的像素类别,我们求助于多 Otsu 阈值处理。
thresholds = ski.filters.threshold_multiotsu(image, classes=3)
regions = np.digitize(image, bins=thresholds)
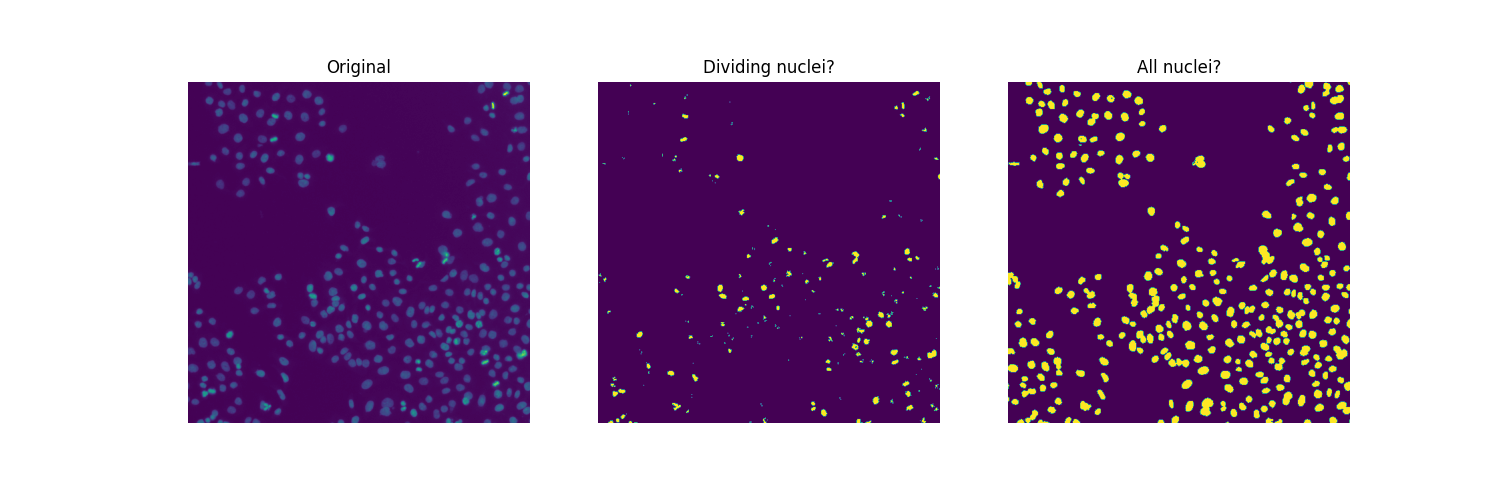
fig, ax = plt.subplots(ncols=2, figsize=(10, 5))
ax[0].imshow(image)
ax[0].set_title('Original')
ax[0].set_axis_off()
ax[1].imshow(regions)
ax[1].set_title('Multi-Otsu thresholding')
ax[1].set_axis_off()
plt.show()

由于存在重叠的细胞核,因此阈值处理不足以分割所有细胞核。如果可以的话,我们可以很容易地计算出此样本的有丝分裂指数
cells = image > thresholds[0]
dividing = image > thresholds[1]
labeled_cells = ski.measure.label(cells)
labeled_dividing = ski.measure.label(dividing)
naive_mi = labeled_dividing.max() / labeled_cells.max()
print(naive_mi)
0.7847222222222222
哇,这不可能! 分裂细胞核的数量
print(labeled_dividing.max())
226
被高估了,而细胞总数
print(labeled_cells.max())
288
被低估了。
计数分裂的细胞核#
显然,中间图中并非所有连接的区域都是分裂的细胞核。一方面,第二个阈值(thresholds[1] 的值)似乎太低,无法将与分裂细胞核相对应的非常亮的区域与许多细胞核中存在的相对较亮的像素分开。另一方面,我们希望获得更平滑的图像,去除小的杂散物体,并可能合并相邻物体的簇(有些可能对应于从一次细胞分裂中出现的两个细胞核)。在某种程度上,我们面临的分裂细胞核的分割挑战与(接触的)细胞的分割挑战相反。
为了找到阈值和滤波参数的合适值,我们通过二分法,在视觉上和手动进行。
higher_threshold = 125
dividing = image > higher_threshold
smoother_dividing = ski.filters.rank.mean(
ski.util.img_as_ubyte(dividing), ski.morphology.disk(4)
)
binary_smoother_dividing = smoother_dividing > 20
fig, ax = plt.subplots(figsize=(5, 5))
ax.imshow(binary_smoother_dividing)
ax.set_title('Dividing nuclei')
ax.set_axis_off()
plt.show()
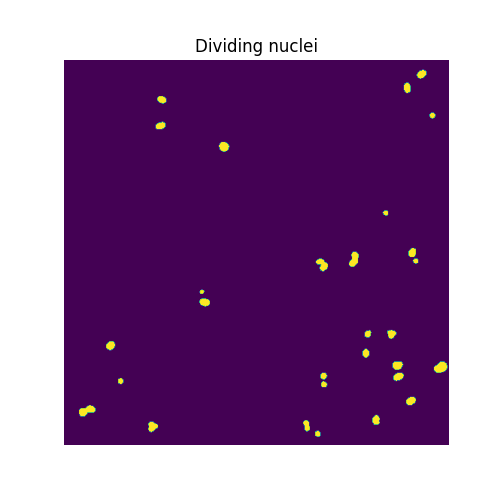
我们剩下
cleaned_dividing = ski.measure.label(binary_smoother_dividing)
print(cleaned_dividing.max())
29
此样本中的分裂细胞核。
分割细胞核#
为了分离重叠的细胞核,我们求助于分水岭分割。 该算法的思想是找到分水岭盆地,就好像从一组 markers 淹没一样。我们将这些标记生成为到背景的距离函数的局部最大值。鉴于细胞核的典型大小,我们传递 min_distance=7,以便局部最大值(因此标记)将至少彼此相距 7 个像素。我们还使用 exclude_border=False,以便包括所有接触图像边界的细胞核。
distance = ndi.distance_transform_edt(cells)
local_max_coords = ski.feature.peak_local_max(
distance, min_distance=7, exclude_border=False
)
local_max_mask = np.zeros(distance.shape, dtype=bool)
local_max_mask[tuple(local_max_coords.T)] = True
markers = ski.measure.label(local_max_mask)
segmented_cells = ski.segmentation.watershed(-distance, markers, mask=cells)
为了方便地可视化分割,我们使用 color.label2rgb 函数对标记的区域进行颜色编码,并使用参数 bg_label=0 指定背景标签。
fig, ax = plt.subplots(ncols=2, figsize=(10, 5))
ax[0].imshow(cells, cmap='gray')
ax[0].set_title('Overlapping nuclei')
ax[0].set_axis_off()
ax[1].imshow(ski.color.label2rgb(segmented_cells, bg_label=0))
ax[1].set_title('Segmented nuclei')
ax[1].set_axis_off()
plt.show()
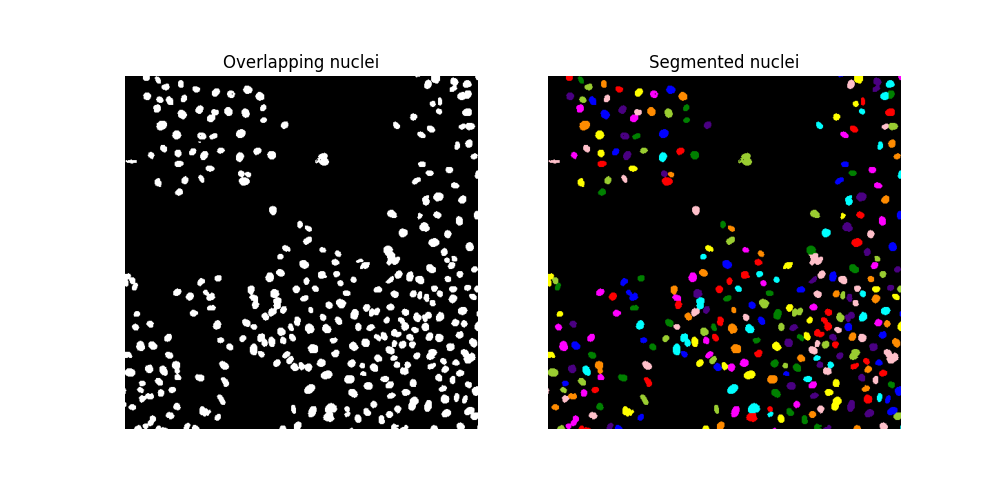
确保分水岭算法已导致识别出更多的细胞核
assert segmented_cells.max() > labeled_cells.max()
最后,我们发现总共有
print(segmented_cells.max())
317
此样本中的细胞。 因此,我们估计有丝分裂指数为
print(cleaned_dividing.max() / segmented_cells.max())
0.0914826498422713
脚本的总运行时间: (0 分钟 3.625 秒)
