注意
转到末尾下载完整示例代码。或通过 Binder 在浏览器中运行此示例
骨骼化#
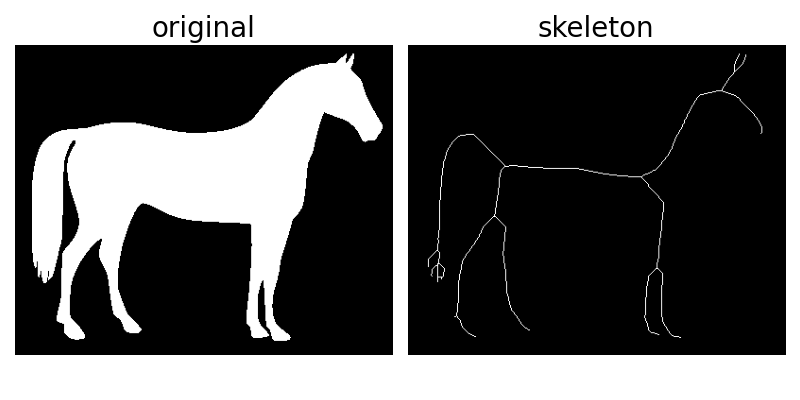
骨骼化将二值对象简化为 1 像素宽的表示。这对于特征提取和/或表示对象的拓扑结构很有用。
skeletonize 通过连续遍历图像来工作。在每次遍历中,识别并移除边界像素,前提是它们不会破坏相应对象的连通性。
from skimage.morphology import skeletonize
from skimage import data
import matplotlib.pyplot as plt
from skimage.util import invert
# Invert the horse image
image = invert(data.horse())
# perform skeletonization
skeleton = skeletonize(image)
# display results
fig, axes = plt.subplots(nrows=1, ncols=2, figsize=(8, 4), sharex=True, sharey=True)
ax = axes.ravel()
ax[0].imshow(image, cmap=plt.cm.gray)
ax[0].axis('off')
ax[0].set_title('original', fontsize=20)
ax[1].imshow(skeleton, cmap=plt.cm.gray)
ax[1].axis('off')
ax[1].set_title('skeleton', fontsize=20)
fig.tight_layout()
plt.show()
张氏方法 vs 李氏方法
skeletonize [Zha84] 通过连续遍历图像来工作,删除对象边界上的像素。这种情况一直持续到不能再删除像素为止。图像与一个掩模相关联,该掩模为每个像素分配一个范围为 [0…255] 的数字,对应于其 8 个相邻像素的每个可能模式。然后使用查找表为像素分配值 0、1、2 或 3,这些值在迭代期间被选择性地删除。
skeletonize(..., method='lee') [Lee94] 使用八叉树数据结构来检查像素的 3x3x3 邻域。该算法通过迭代扫描图像来工作,并在每次迭代中删除像素,直到图像停止变化。每次迭代都包含两个步骤:首先,组装一个要删除的候选像素列表;然后,按顺序重新检查此列表中的像素,以更好地保留图像的连通性。
请注意,李氏方法 [Lee94] 设计用于 3D 图像,并为 3D 图像自动选择。为了说明目的,我们将此算法应用于 2D 图像。
用于细化数字模式的快速并行算法,T. Y. Zhang 和 C. Y. Suen,ACM 通讯,1984 年 3 月,第 27 卷,第 3 期。
import matplotlib.pyplot as plt
from skimage.morphology import skeletonize
blobs = data.binary_blobs(200, blob_size_fraction=0.2, volume_fraction=0.35, rng=1)
skeleton = skeletonize(blobs)
skeleton_lee = skeletonize(blobs, method='lee')
fig, axes = plt.subplots(1, 3, figsize=(8, 4), sharex=True, sharey=True)
ax = axes.ravel()
ax[0].imshow(blobs, cmap=plt.cm.gray)
ax[0].set_title('original')
ax[0].axis('off')
ax[1].imshow(skeleton, cmap=plt.cm.gray)
ax[1].set_title('skeletonize')
ax[1].axis('off')
ax[2].imshow(skeleton_lee, cmap=plt.cm.gray)
ax[2].set_title('skeletonize (Lee 94)')
ax[2].axis('off')
fig.tight_layout()
plt.show()

中轴骨骼化
对象的中轴是对象边界上具有多个最近点的所有点的集合。它通常被称为拓扑骨骼,因为它是对象的 1 像素宽的骨骼,与原始对象具有相同的连通性。
这里,我们使用中轴变换来计算前景对象的宽度。由于函数 medial_axis 除了中轴(使用关键字参数 return_distance=True)之外还返回距离变换,因此可以使用此函数计算中轴所有点到背景的距离。这给出了对象局部宽度的估计。
对于分支较少的骨骼,应首选 skeletonize。
from skimage.morphology import medial_axis, skeletonize
# Generate the data
blobs = data.binary_blobs(200, blob_size_fraction=0.2, volume_fraction=0.35, rng=1)
# Compute the medial axis (skeleton) and the distance transform
skel, distance = medial_axis(blobs, return_distance=True)
# Compare with other skeletonization algorithms
skeleton = skeletonize(blobs)
skeleton_lee = skeletonize(blobs, method='lee')
# Distance to the background for pixels of the skeleton
dist_on_skel = distance * skel
fig, axes = plt.subplots(2, 2, figsize=(8, 8), sharex=True, sharey=True)
ax = axes.ravel()
ax[0].imshow(blobs, cmap=plt.cm.gray)
ax[0].set_title('original')
ax[0].axis('off')
ax[1].imshow(dist_on_skel, cmap='magma')
ax[1].contour(blobs, [0.5], colors='w')
ax[1].set_title('medial_axis')
ax[1].axis('off')
ax[2].imshow(skeleton, cmap=plt.cm.gray)
ax[2].set_title('skeletonize')
ax[2].axis('off')
ax[3].imshow(skeleton_lee, cmap=plt.cm.gray)
ax[3].set_title("skeletonize (Lee 94)")
ax[3].axis('off')
fig.tight_layout()
plt.show()

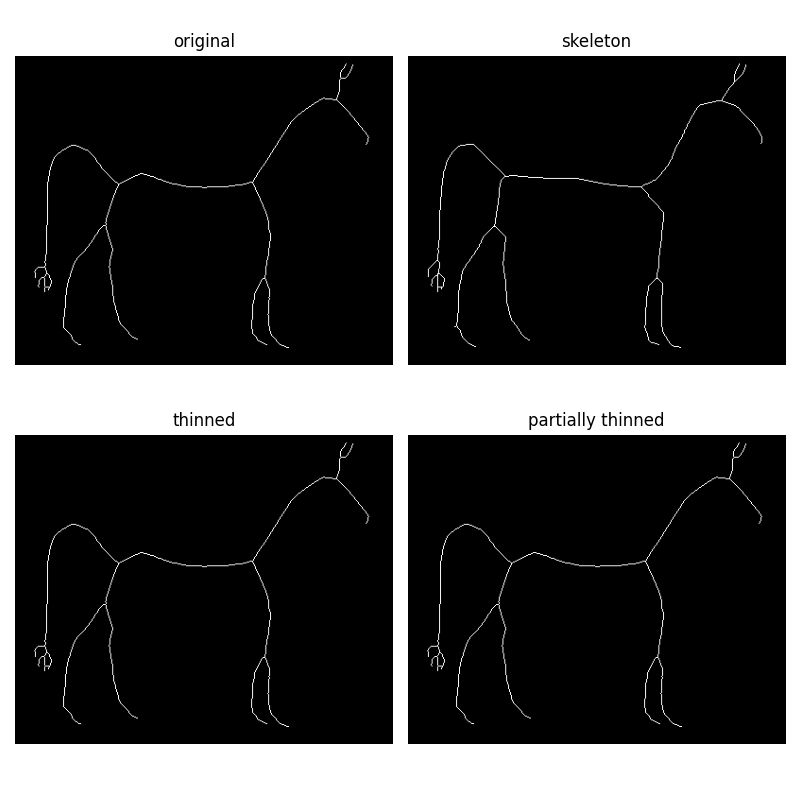
形态学细化
形态学细化(在 thin 函数中实现)的工作原理与 skeletonize 相同:在每次迭代中从边界删除像素,直到不能在不改变连通性的情况下删除像素。不同的删除规则可以加快骨骼化速度,并导致不同的最终骨骼。
thin 函数还采用可选的 max_num_iter 关键字参数来限制细化迭代的次数,从而产生相对较粗的骨骼。
from skimage.morphology import skeletonize, thin
skeleton = skeletonize(image)
thinned = thin(image)
thinned_partial = thin(image, max_num_iter=25)
fig, axes = plt.subplots(2, 2, figsize=(8, 8), sharex=True, sharey=True)
ax = axes.ravel()
ax[0].imshow(image, cmap=plt.cm.gray)
ax[0].set_title('original')
ax[0].axis('off')
ax[1].imshow(skeleton, cmap=plt.cm.gray)
ax[1].set_title('skeleton')
ax[1].axis('off')
ax[2].imshow(thinned, cmap=plt.cm.gray)
ax[2].set_title('thinned')
ax[2].axis('off')
ax[3].imshow(thinned_partial, cmap=plt.cm.gray)
ax[3].set_title('partially thinned')
ax[3].axis('off')
fig.tight_layout()
plt.show()
脚本的总运行时间:(0 分钟 2.332 秒)
