注意
转到末尾下载完整的示例代码。 或通过 Binder 在您的浏览器中运行此示例
探索 3D 图像(细胞)#
本教程是对三维图像处理的介绍。 有关 3D 数据集的快速入门,请参阅具有 3 个或更多空间维度的数据集。 图像表示为 numpy 数组。 单通道或灰度图像是一个形状为 (n_row, n_col) 的像素强度 2D 矩阵,其中 n_row(resp. n_col)表示 行(resp. 列)的数量。 我们可以将 3D 体积构造为一系列 2D 平面,使 3D 图像的形状为 (n_plane, n_row, n_col),其中 n_plane 是平面的数量。 多通道或 RGB(A) 图像在最终位置有一个额外的 通道 维度,其中包含颜色信息。
这些约定在下表中进行了总结
图像类型 |
坐标 |
|---|---|
2D 灰度 |
|
2D 多通道 |
|
3D 灰度 |
|
3D 多通道 |
|
一些 3D 图像是在每个维度上具有相等分辨率的情况下构造的(例如,同步加速器断层扫描或球体的计算机生成渲染)。但是大多数实验数据是在三个维度之一中以较低的分辨率捕获的,例如,拍摄薄片以将 3D 结构近似为 2D 图像的堆栈。每个维度中像素之间的距离(称为间距)被编码为一个元组,并被一些 skimage 函数接受为参数,并且可以用于调整对滤波器的贡献。
本教程中使用的数据由艾伦细胞科学研究所提供。它们在 行 和 列 维度上被下采样了 4 倍,以减小它们的大小,从而减少计算时间。间距信息由用于对细胞成像的显微镜报告。
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d.art3d import Poly3DCollection
import numpy as np
import plotly
import plotly.express as px
from skimage import exposure, util
from skimage.data import cells3d
加载和显示 3D 图像#
data = util.img_as_float(cells3d()[:, 1, :, :]) # grab just the nuclei
print(f'shape: {data.shape}')
print(f'dtype: {data.dtype}')
print(f'range: ({data.min()}, {data.max()})')
# Report spacing from microscope
original_spacing = np.array([0.2900000, 0.0650000, 0.0650000])
# Account for downsampling of slices by 4
rescaled_spacing = original_spacing * [1, 4, 4]
# Normalize spacing so that pixels are a distance of 1 apart
spacing = rescaled_spacing / rescaled_spacing[2]
print(f'microscope spacing: {original_spacing}\n')
print(f'rescaled spacing: {rescaled_spacing} (after downsampling)\n')
print(f'normalized spacing: {spacing}\n')
shape: (60, 256, 256)
dtype: float64
range: (0.0, 1.0)
microscope spacing: [0.29 0.065 0.065]
rescaled spacing: [0.29 0.26 0.26] (after downsampling)
normalized spacing: [1.11538462 1. 1. ]
让我们尝试可视化我们的 3D 图像。不幸的是,许多图像查看器(例如 matplotlib 的 imshow)只能显示 2D 数据。我们可以看到,当我们尝试查看 3D 数据时,它们会引发错误
Invalid shape (60, 256, 256) for image data
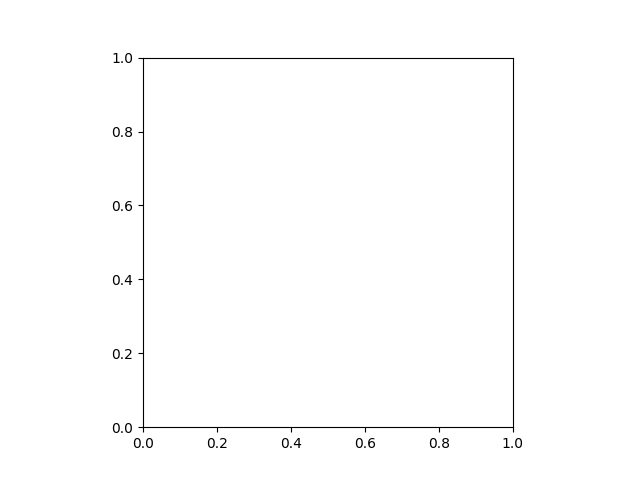
imshow 函数只能显示灰度和 RGB(A) 2D 图像。因此,我们可以使用它来可视化 2D 平面。通过固定一个轴,我们可以观察到图像的三个不同视图。
def show_plane(ax, plane, cmap="gray", title=None):
ax.imshow(plane, cmap=cmap)
ax.set_axis_off()
if title:
ax.set_title(title)
(n_plane, n_row, n_col) = data.shape
_, (a, b, c) = plt.subplots(ncols=3, figsize=(15, 5))
show_plane(a, data[n_plane // 2], title=f'Plane = {n_plane // 2}')
show_plane(b, data[:, n_row // 2, :], title=f'Row = {n_row // 2}')
show_plane(c, data[:, :, n_col // 2], title=f'Column = {n_col // 2}')
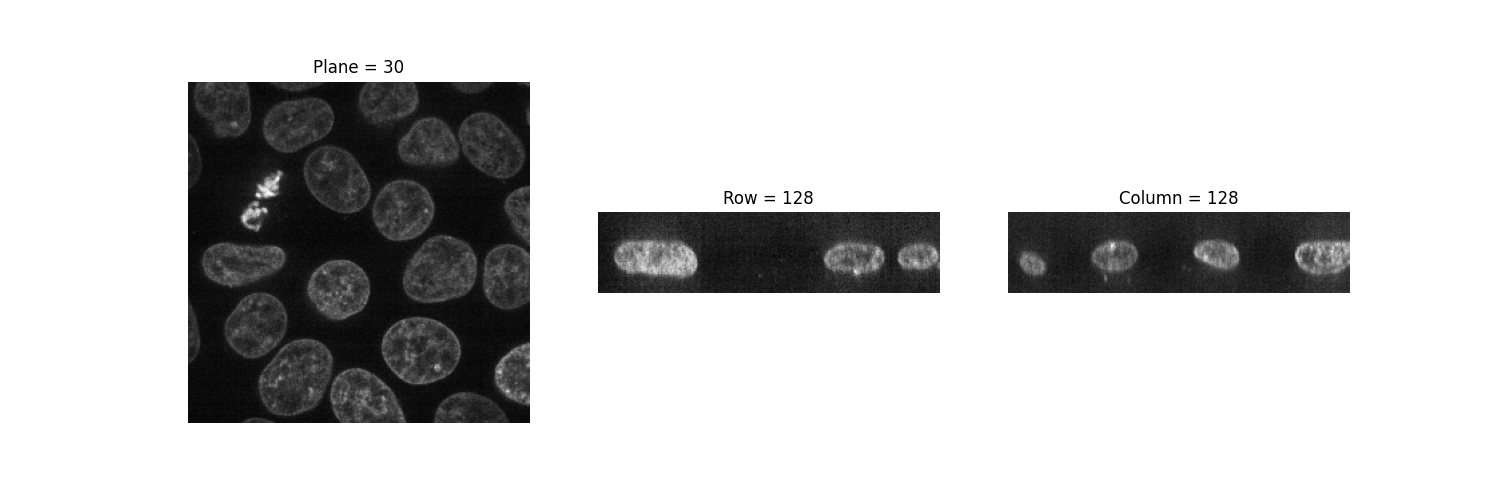
如前所述,三维图像可以看作是一系列二维平面。让我们编写一个辅助函数 display,以创建多个平面的蒙太奇。默认情况下,显示每隔一个平面。
def display(im3d, cmap='gray', step=2):
data_montage = util.montage(im3d[::step], padding_width=4, fill=np.nan)
_, ax = plt.subplots(figsize=(16, 14))
ax.imshow(data_montage, cmap=cmap)
ax.set_axis_off()
display(data)

或者,我们可以使用 Jupyter 小部件以交互方式探索这些平面(切片)。让用户选择要显示的切片,并显示此切片在 3D 数据集中的位置。请注意,您无法在静态 HTML 页面中看到 Jupyter 小部件的工作方式,就像此示例的在线版本一样。要使以下代码正常工作,您需要一个本地或云端运行的 Jupyter 内核:请参阅此页面底部,以下载 Jupyter 笔记本并在您的计算机上运行它,或直接在 Binder 中打开它。除了活动内核之外,您还需要一个 Web 浏览器:在纯 Python 中运行代码也行不通。
def slice_in_3D(ax, i):
# From https://stackoverflow.com/questions/44881885/python-draw-3d-cube
Z = np.array(
[
[0, 0, 0],
[1, 0, 0],
[1, 1, 0],
[0, 1, 0],
[0, 0, 1],
[1, 0, 1],
[1, 1, 1],
[0, 1, 1],
]
)
Z = Z * data.shape
r = [-1, 1]
X, Y = np.meshgrid(r, r)
# Plot vertices
ax.scatter3D(Z[:, 0], Z[:, 1], Z[:, 2])
# List sides' polygons of figure
verts = [
[Z[0], Z[1], Z[2], Z[3]],
[Z[4], Z[5], Z[6], Z[7]],
[Z[0], Z[1], Z[5], Z[4]],
[Z[2], Z[3], Z[7], Z[6]],
[Z[1], Z[2], Z[6], Z[5]],
[Z[4], Z[7], Z[3], Z[0]],
[Z[2], Z[3], Z[7], Z[6]],
]
# Plot sides
ax.add_collection3d(
Poly3DCollection(
verts, facecolors=(0, 1, 1, 0.25), linewidths=1, edgecolors="darkblue"
)
)
verts = np.array([[[0, 0, 0], [0, 0, 1], [0, 1, 1], [0, 1, 0]]])
verts = verts * (60, 256, 256)
verts += [i, 0, 0]
ax.add_collection3d(
Poly3DCollection(verts, facecolors="magenta", linewidths=1, edgecolors="black")
)
ax.set_xlabel("plane")
ax.set_xlim(0, 100)
ax.set_ylabel("row")
ax.set_zlabel("col")
# Autoscale plot axes
scaling = np.array([getattr(ax, f'get_{dim}lim')() for dim in "xyz"])
ax.auto_scale_xyz(*[[np.min(scaling), np.max(scaling)]] * 3)
def explore_slices(data, cmap="gray"):
from ipywidgets import interact
N = len(data)
@interact(plane=(0, N - 1))
def display_slice(plane=34):
fig, ax = plt.subplots(figsize=(20, 5))
ax_3D = fig.add_subplot(133, projection="3d")
show_plane(ax, data[plane], title=f'Plane {plane}', cmap=cmap)
slice_in_3D(ax_3D, plane)
plt.show()
return display_slice
explore_slices(data)
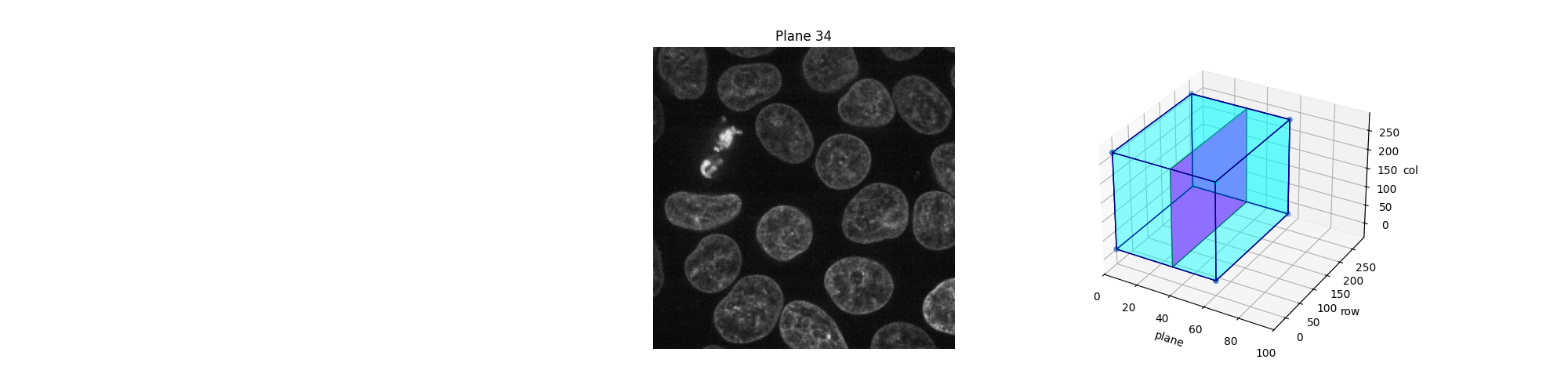
interactive(children=(IntSlider(value=34, description='plane', max=59), Output()), _dom_classes=('widget-interact',))
<function explore_slices.<locals>.display_slice at 0x7f287ebaa020>
调整曝光#
Scikit-image 的 exposure 模块包含许多用于调整图像对比度的函数。这些函数对像素值进行操作。通常,不需要考虑图像维度或像素间距。但是,在局部曝光校正中,可能需要调整窗口大小,以确保沿每个轴的实际坐标中的大小相等。
伽马校正可以使图像变亮或变暗。它将一个幂律变换应用于图像中的每个像素,其中 gamma 表示幂律指数:gamma < 1 会使图像变亮,而 gamma > 1 会使图像变暗。
def plot_hist(ax, data, title=None):
# Helper function for plotting histograms
ax.hist(data.ravel(), bins=256)
ax.ticklabel_format(axis="y", style="scientific", scilimits=(0, 0))
if title:
ax.set_title(title)
gamma_low_val = 0.5
gamma_low = exposure.adjust_gamma(data, gamma=gamma_low_val)
gamma_high_val = 1.5
gamma_high = exposure.adjust_gamma(data, gamma=gamma_high_val)
_, ((a, b, c), (d, e, f)) = plt.subplots(nrows=2, ncols=3, figsize=(12, 8))
show_plane(a, data[32], title='Original')
show_plane(b, gamma_low[32], title=f'Gamma = {gamma_low_val}')
show_plane(c, gamma_high[32], title=f'Gamma = {gamma_high_val}')
plot_hist(d, data)
plot_hist(e, gamma_low)
plot_hist(f, gamma_high)
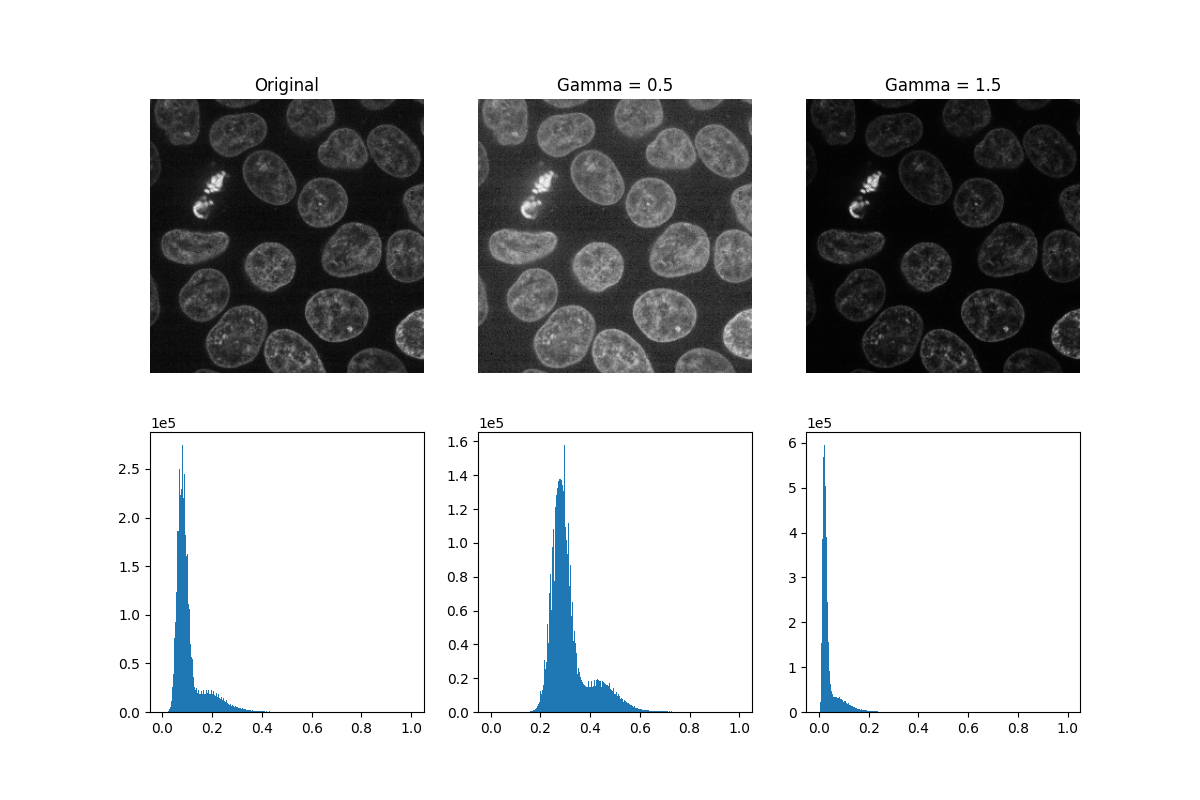
直方图均衡化通过重新分配像素强度来提高图像的对比度。最常见的像素强度被分散开来,从而增加低对比度区域的对比度。这种方法的一个缺点是可能会增强背景噪声。
equalized_data = exposure.equalize_hist(data)
display(equalized_data)

和之前一样,如果我们有一个正在运行的 Jupyter 内核,我们可以交互式地探索上述切片。
explore_slices(equalized_data)
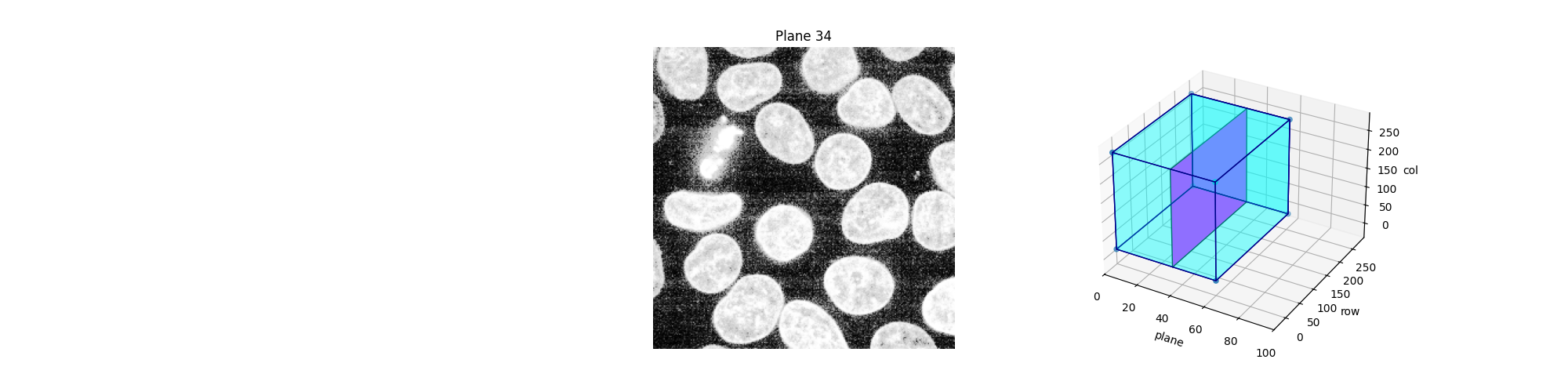
interactive(children=(IntSlider(value=34, description='plane', max=59), Output()), _dom_classes=('widget-interact',))
<function explore_slices.<locals>.display_slice at 0x7f287efc22a0>
现在让我们绘制直方图均衡化前后的图像直方图。下面,我们绘制了相应的累积分布函数 (CDF)。
_, ((a, b), (c, d)) = plt.subplots(nrows=2, ncols=2, figsize=(16, 8))
plot_hist(a, data, title="Original histogram")
plot_hist(b, equalized_data, title="Equalized histogram")
cdf, bins = exposure.cumulative_distribution(data.ravel())
c.plot(bins, cdf, "r")
c.set_title("Original CDF")
cdf, bins = exposure.cumulative_distribution(equalized_data.ravel())
d.plot(bins, cdf, "r")
d.set_title("Histogram equalization CDF")
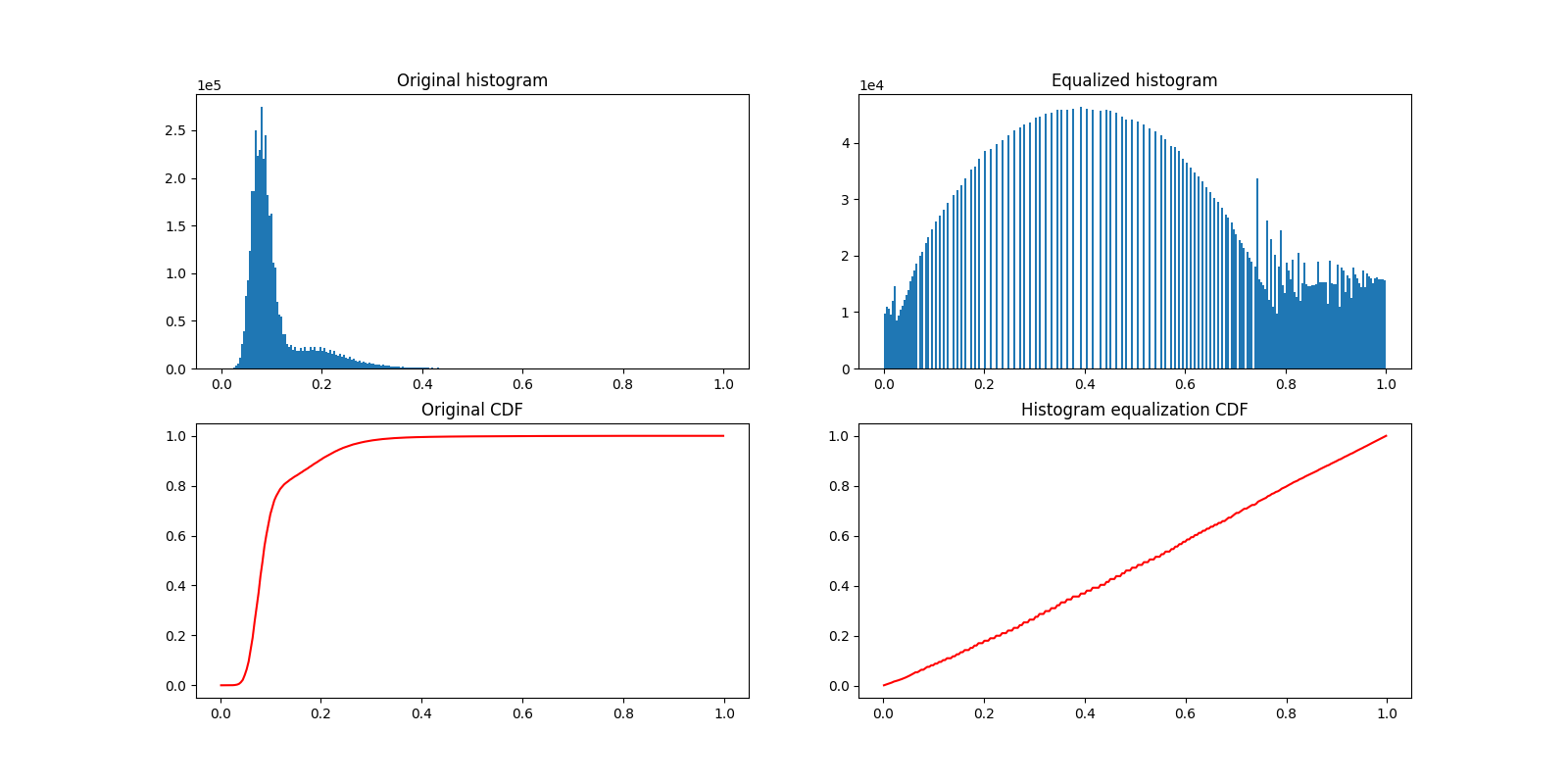
Text(0.5, 1.0, 'Histogram equalization CDF')
大多数实验图像都受到椒盐噪声的影响。一些明亮的伪影会降低感兴趣像素的相对强度。一种提高对比度的简单方法是裁剪最低和最高的极端像素值。裁剪最暗和最亮的 0.5% 的像素将增加图像的整体对比度。
vmin, vmax = np.percentile(data, q=(0.5, 99.5))
clipped_data = exposure.rescale_intensity(
data, in_range=(vmin, vmax), out_range=np.float32
)
display(clipped_data)

或者,我们可以使用 Plotly Express 交互式地探索这些平面(切片)。请注意,这可以在静态 HTML 页面中工作!
fig = px.imshow(data, animation_frame=0, binary_string=True)
fig.update_xaxes(showticklabels=False)
fig.update_yaxes(showticklabels=False)
fig.update_layout(autosize=False, width=500, height=500, coloraxis_showscale=False)
# Drop animation buttons
fig['layout'].pop('updatemenus')
plotly.io.show(fig)
plt.show()
脚本总运行时间: (0 分钟 10.688 秒)
